新建
首页
探究
订阅
期刊库
学者
知识库
Notebooks
课程
Apps
比赛
实验室
Uni-Lab
计算
任务
文件
更多
节点
数据集
镜像
项目
数据库
CARSI权益
多端下载
中文
登录
APP介绍
使用手册
讨论区2
App 介绍
分子对接是一种通过计算机模拟快速评估候选分子与蛋白质受体结合亲和力的方法。由于其高通量、快速和良好的富集能力,它在药物设计的早期阶段被广泛用于虚拟筛选,以在大规模分子库中寻找潜在的活性分子。
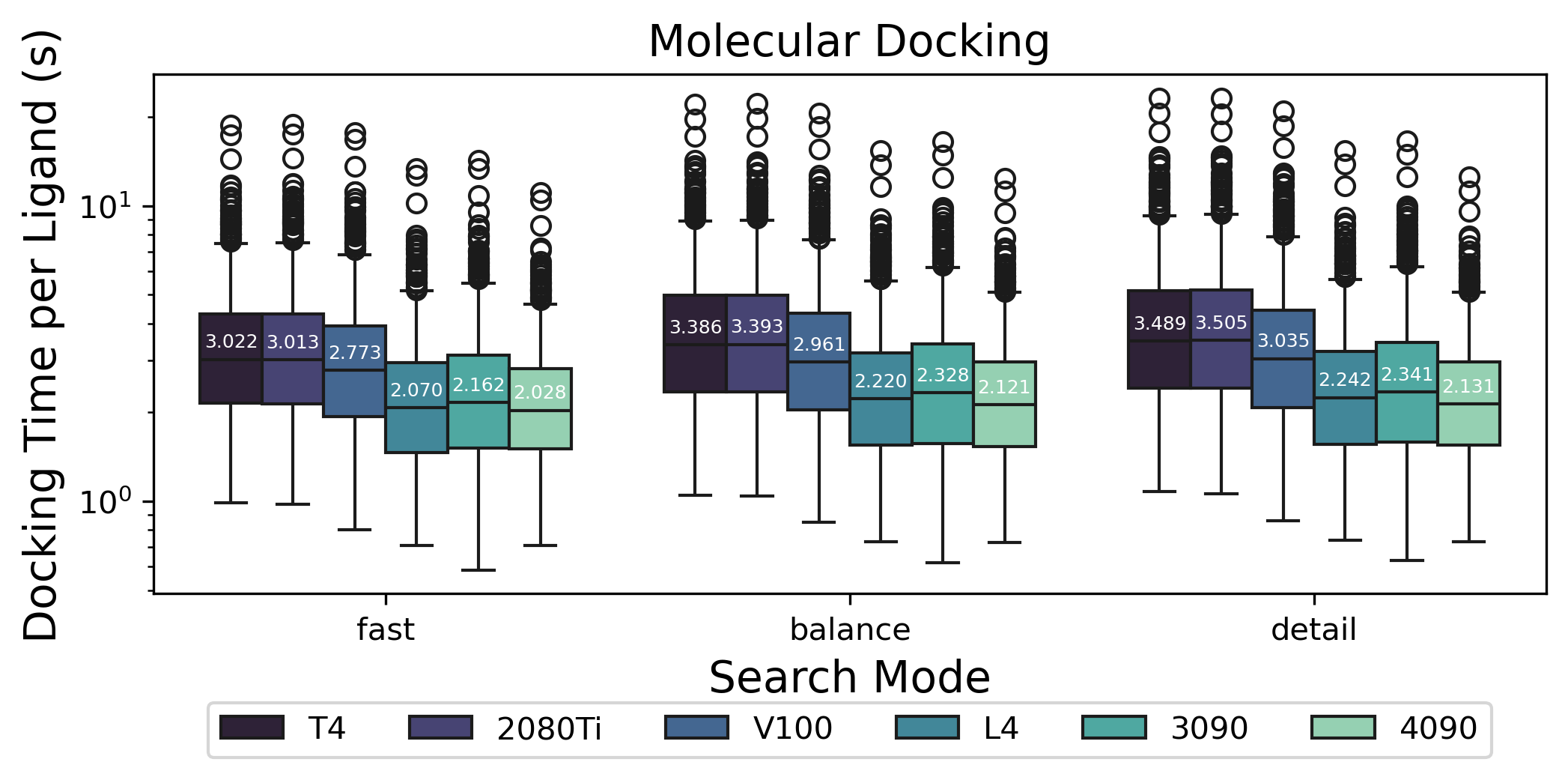
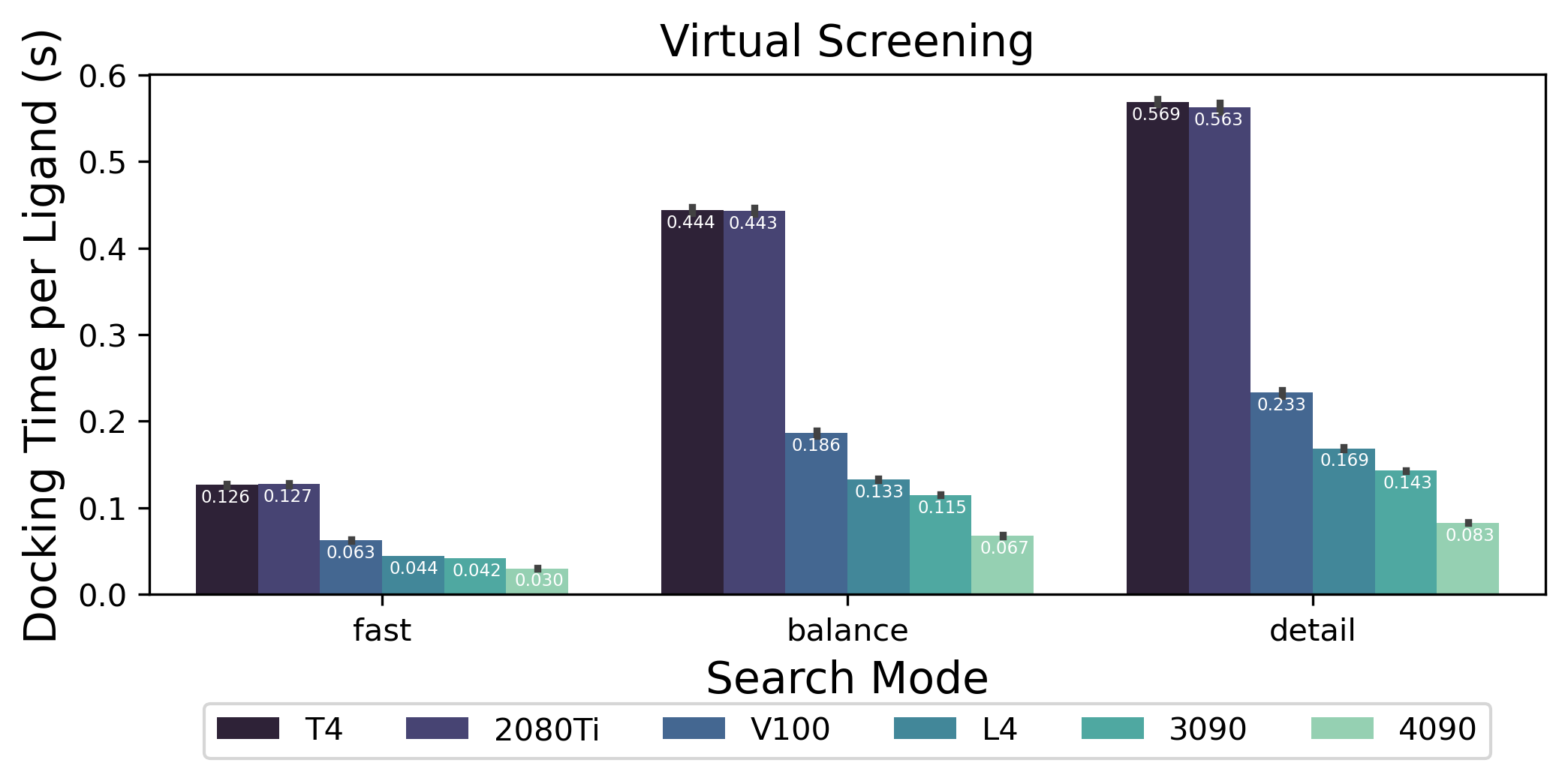
由DPTechnology开发的Uni-Dock是一个GPU加速的分子对接引擎,可以在GPU上以超过单个CPU 3000倍的速度完成虚拟筛选任务。2023年6月,Uni-Dock作为封面文章登上了《化学理论与计算》杂志[1]。在最新版本Uni-Dock v1.1中,虚拟筛选速度可达0.03秒/配体。这意味着使用100个GPU集群,您只需8.3小时就能完成1亿个分子的虚拟筛选。
Uni-Dock是由DeepModeling社区孵化的开源软件,并在LGPL许可下可用。任何人都可以在GitHub上访问Uni-Dock的最新版本。
这个应用程序基于最新版本的Uni-Dock,并旨在简化安装和部署过程,同时利用Bohrium的大量计算资源提供端到端的快速虚拟筛选服务。
下面展示了Uni-Dock进行单配体分子对接和多配体虚拟筛选的速度:
最佳实践(optional)
Example 1
-
Inputs:
-
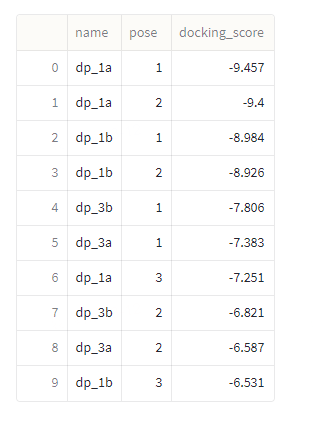
Outputs(For reference only, the scores may not be the same):
- Result files in outputs folder.
- Score table:
参考文献
[1] Yu Y, Cai C, Wang J, et al. Uni-dock: Gpu-accelerated docking enables ultralarge virtual screening[J]. Journal of chemical theory and computation, 2023, 19(11): 3336-3345.
引用格式
Uni-Dock
@article{yu2023uni, title={Uni-dock: Gpu-accelerated docking enables ultralarge virtual screening}, author={Yu, Yuejiang and Cai, Chun and Wang, Jiayue and Bo, Zonghua and Zhu, Zhengdan and Zheng, Hang}, journal={Journal of chemical theory and computation}, volume={19}, number={11}, pages={3336--3345}, year={2023}, publisher={ACS Publications} }
推荐App
Uni-Fold
高精度蛋白质结构预测工具,支持蛋白复合物、同源高聚体预测Uni-View
Uni-View:一个具有编辑、测量、样式定制和图像导出功能的分子可视化工具。Uni-Mol Docking V2
使用Uni-Mol Docking V2进行分子-蛋白配体复合物结构预测。RNA Secondary Structure Prediction
预训练模型与热力学参数优化的高精度RNA二级结构预测工具Uni-Dock
Uni-Dock, a GPU-accelerated molecular docking program that supports various scoring functions including vina, vinardo, and ad4.PTM Predictor
蛋白质翻译后修饰位点预测 (PTM Predictor) 是一个基于深度学习精确预测蛋白质多种翻译后修饰位点的全面工具。推荐文章
从蛋白/小分子准备开始的Uni-Dock分子对接全流程介绍yuanyn

更新于 2023-07-10
3 赞8 转存文件
yuyj_depart@dp.tech

更新于 2023-07-30
2 赞1 转存文件

郑行

更新于 2023-06-18
14 赞36 转存文件
yuanyn

更新于 2024-02-28
1 赞8 转存文件

zhougm@dp.tech

更新于 2023-08-31
2 转存文件

Zhifeng Gao

更新于 2023-09-14
1 赞1 转存文件