新建
3.2_蛋白多聚体(复合物)结构预测

songk@dp.tech

推荐镜像 :Basic Image:ubuntu22.04-py3.10
推荐机型 :c2_m4_cpu
赞
1
目录
数据集
师资培训(v5)
基于Uni-Fold的蛋白多聚体结构预测与评估
导言
Hermite®平台中的Uni-Fold模块,首次成功复现了AlphaFold2的全规模训练,可实现蛋白质单体及蛋白多聚体的结构预测。在本教程中,您将学习使用Uni-Fold模块实现蛋白质多聚体的结构预测,并通过调用Hermite中的Protein Alignment模块,进行预测结构与真实实验结构的比对,评价蛋白质结构预测的准确性。
获取Protein Sequence:
- 从RCSB网站直接下载。
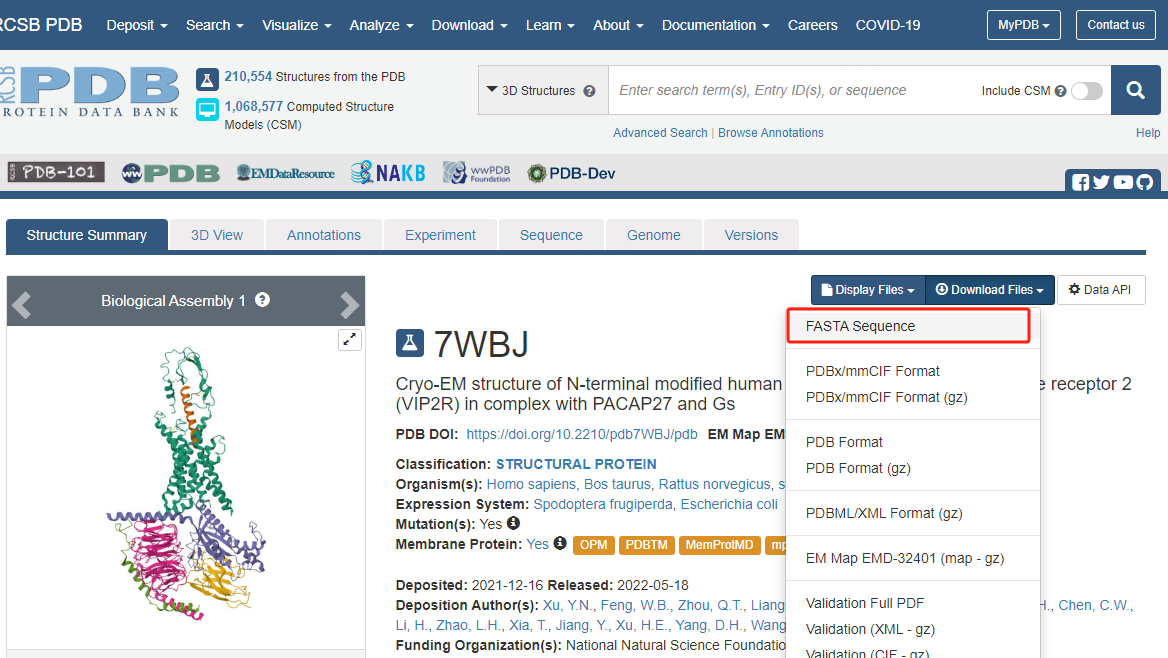
- 已下载好的文件:rcsb_pdb_7BWJ.fasta
1. Protein Folding
1.1 入口
- 左侧通用菜单栏Function → Structure Modeling → Protein Folding。
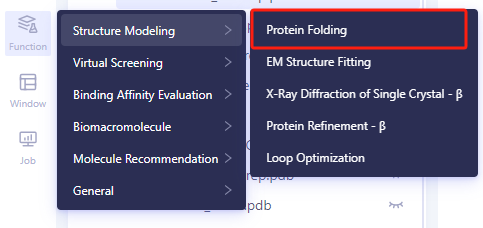
1.2 蛋白序列导入
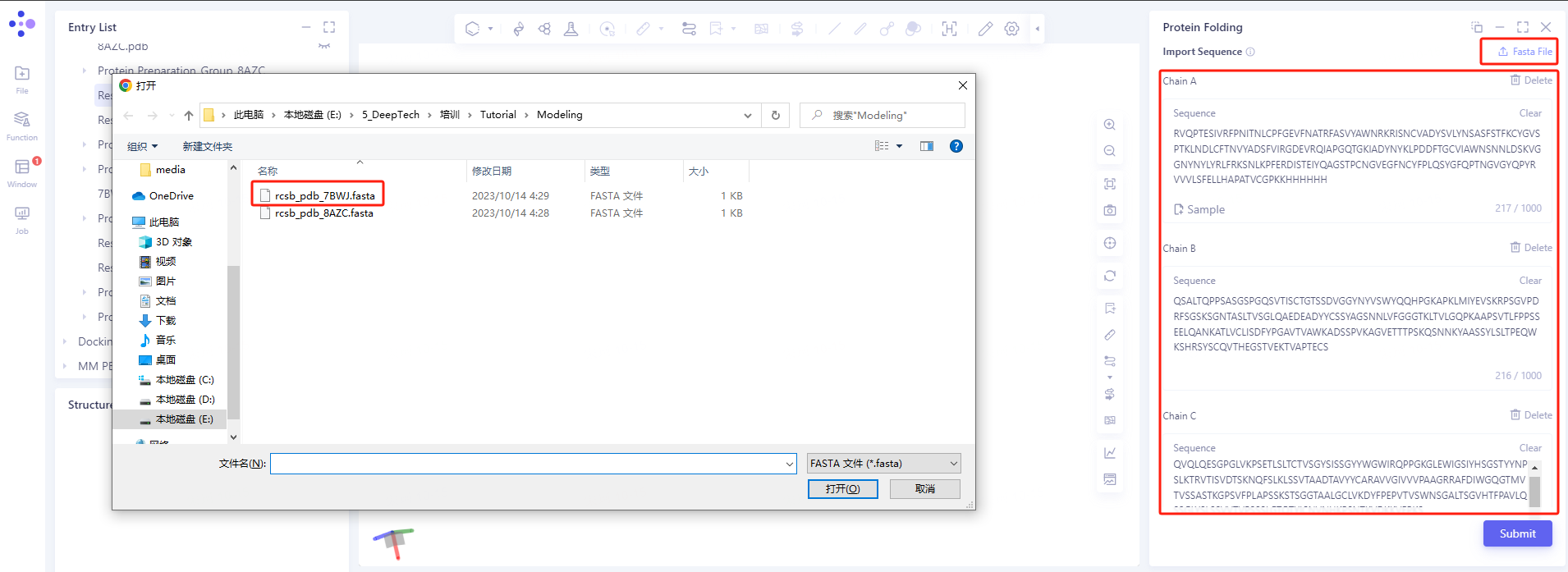
在右侧出现的Protein Folding界面(红框内所示)中上传蛋白序列的fasta文件。
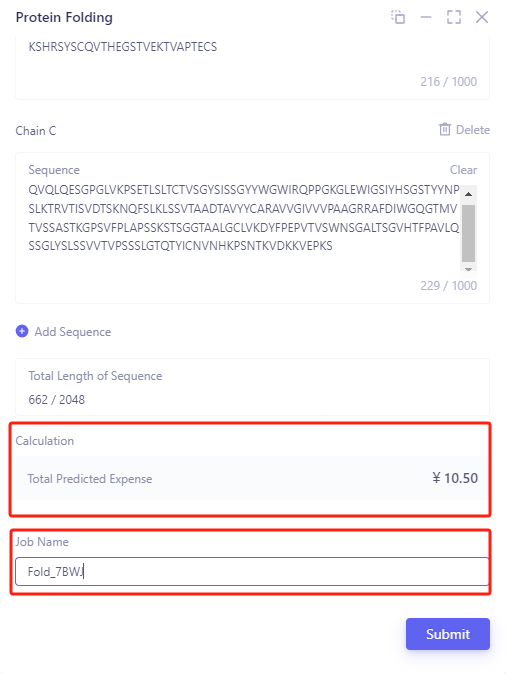
在“Calculation”栏,显示了预估的计算Balance消耗。
Job Name处将该任务进行命名为 Fold_7BWJ 后,点击Submit提交任务。
1.3 结果查看
- 点击左侧通用菜单栏的Job → 在弹出的Job List界面处找出 Fold_7BWJ 任务 → 点击 Show。
蛋白预测结果显示于 3D Workspace 界面中。同时预测结构的置信度评价(pTM、ipTM和model confidence,分数越高,预测结构的置信度越高)和PAE分析(Predicted Aligned Error,数值越低,相应氨基酸位置的预测误差越小)也会显示在独立的Web页面。
- pTM:predicted TM-score,估计内在模型精度
- iPTM:Inerface pTM,隶属不同Chain的残基之间的精度
- model confidence = 0.8 · ipTM + 0.2 · pTM
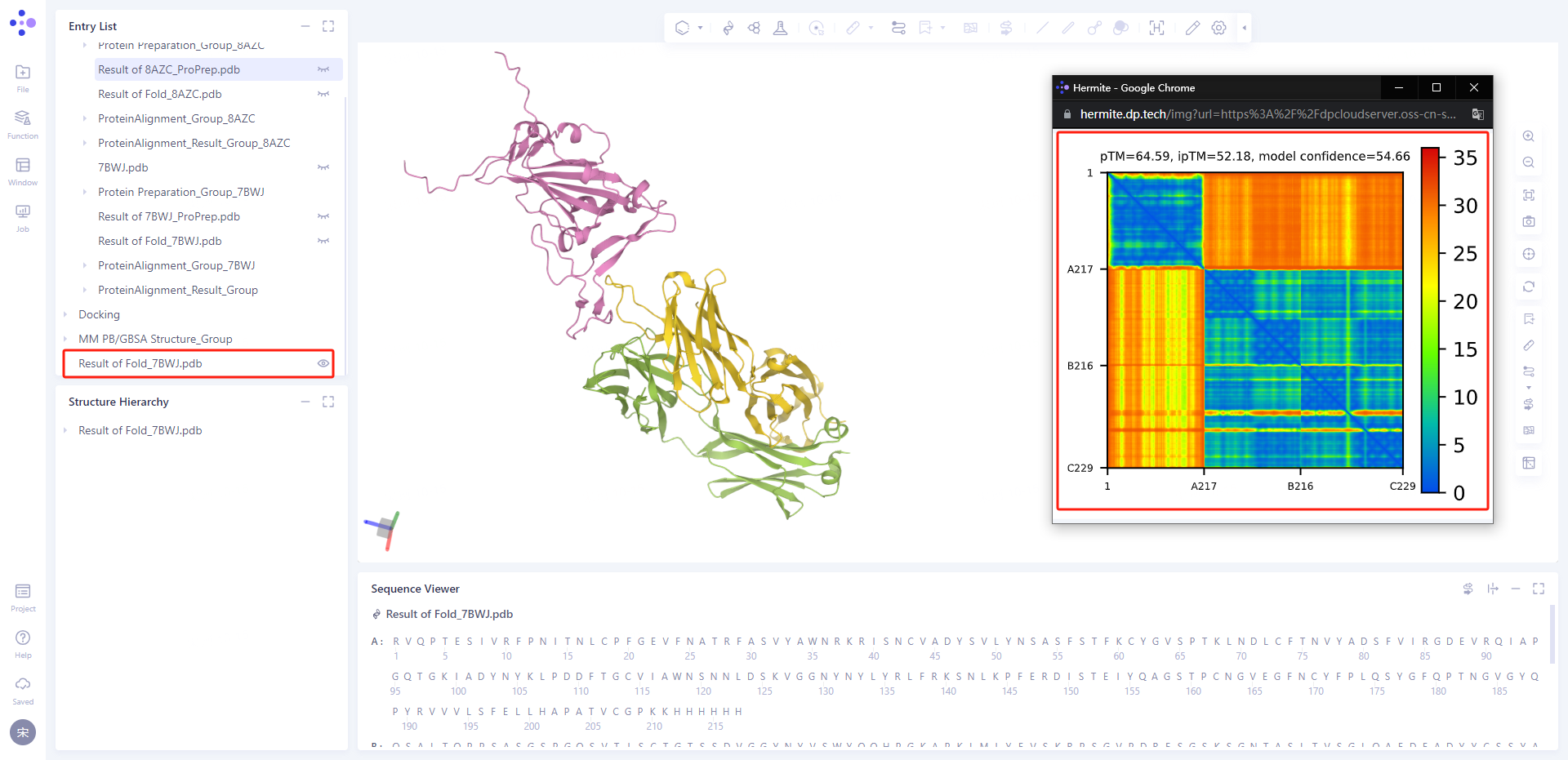
从PAE的分析来看,蛋白复合物预测的结构中,Antigen(Chain A)和Antibody(Chain B和Chain C)内部的误差较小,但是Antigen和Antibody之间的相对位置误差较大。蛋白复合物结构整体的置信度不算非常高(model confidence = 54.66)。这里需要说明的是,抗原抗体复合物的结构预测本身是一个比较复杂和困难的问题。
2. Protein Alignment
2.1 将蛋白结构导入3D Workspace窗口
在同一个 Project中,点击左侧通用菜单栏File → Get PDB。
在弹出的窗口中输入PDB ID:7BWJ,点击Import导入该蛋白质结构。推进使用Protein Preparation模块对导入的Protein进行Preparation。
2.2 调用Protein Alignment模块
2.2.1 入口
- 调用Protein Alignment 模块比对蛋白结构:左侧通用菜单栏Function → General → Protein Alignment。
2.2.2 参考蛋白结构导入
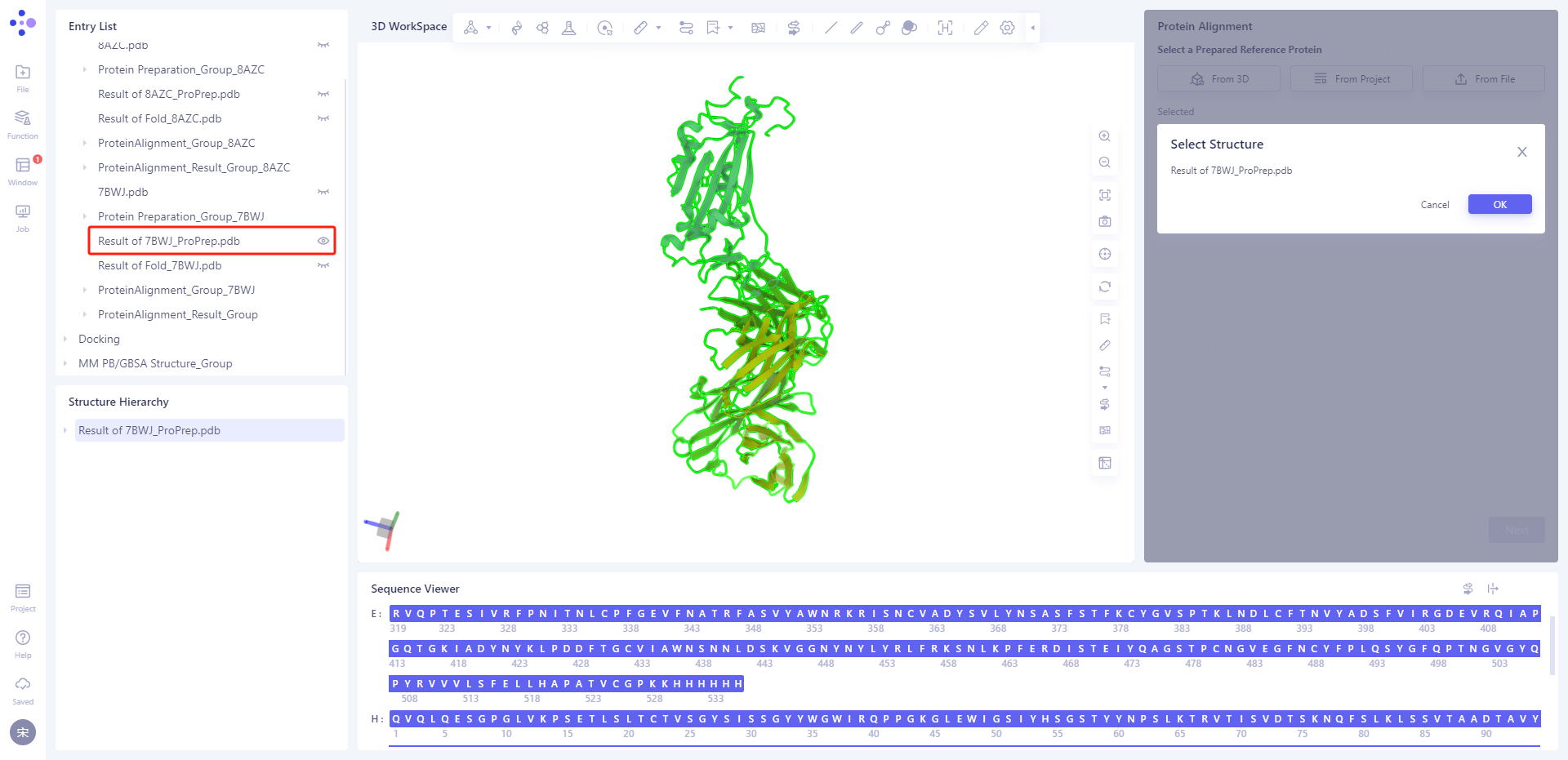
- 界面右侧出现的Protein Alignment操作窗口,首先导入参考蛋白结构:点击Select Structure,弹出Select Structure窗口 → 在Structure Hierarchy内选中Preparation好的7BWJ蛋白结构 → Select Structure窗口点击OK。
2.2.3 需比对蛋白结构导入
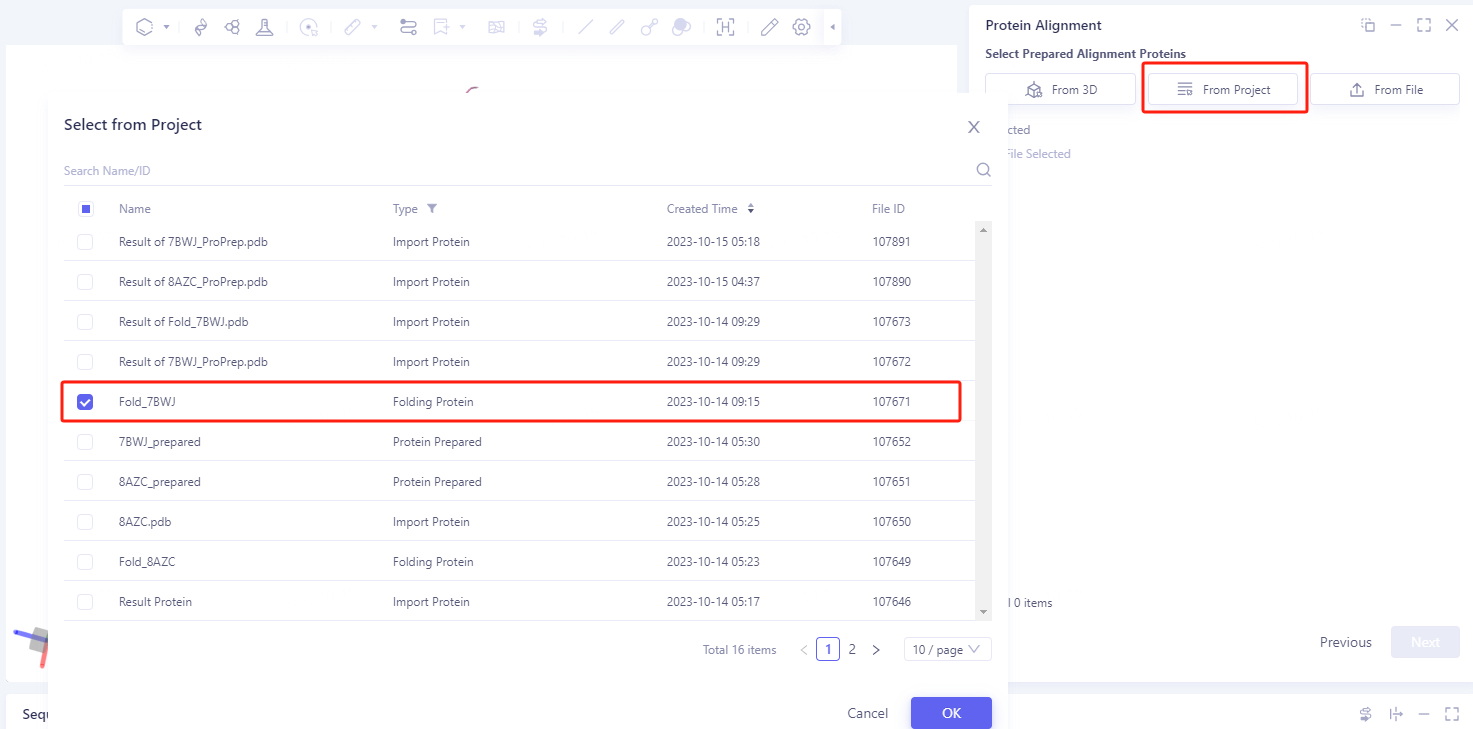
- Next点击后进入比对蛋白的选择窗口,导入需要比对的蛋白结构:点击Select from Project,弹出Select from Project窗口 → 在Select from Project窗口中选择Fold_7BWJ → 点击OK。
2.2.4 参数设置
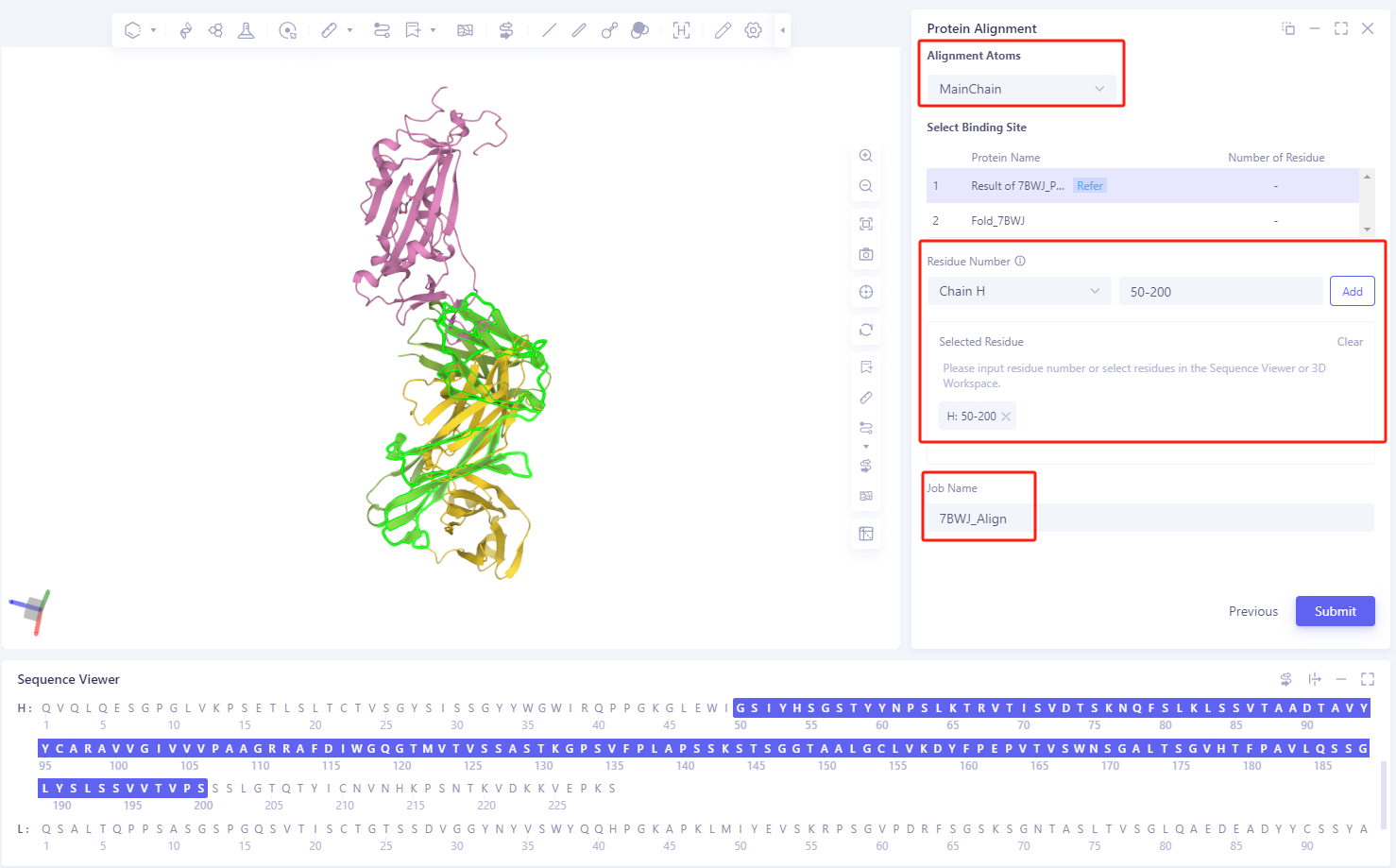
- Next点击后进入参数设置页面,Alignment Atoms选择基于蛋白之间的MainChain进行比对,Residue的范围包括Chain H的ResID 50-200。Job Name处将该任务命名为7BWJ_Align,点击Submit提交该任务。
2.3 结果分析
2.3.1入口
- 左侧通用菜单栏Job → 在弹出的Job List窗口中找到任务 7BWJ_Align → 点击Operation下的操作 Show 展示该任务。
2.3.2结果展示与说明
- 蛋白结构的叠合图像结果显示于3D Workspace窗口,界面右侧出现Protein Alignment Result窗口显示蛋白的比对结果。
- 经过蛋白结构比对,发现Uni-Fold预测的抗原抗体复合物的结构与原蛋白结构(PDB 7BWJ)中抗体结构(Align时选中的Reference)的RMSD值为1.67 Å,相似性较高。但抗原与抗体的相对位置有明显的差异(3D Workspace中左上区域),这与PAE的分析结果一致。
代码
文本
点个赞吧
本文被以下合集收录
蛋白

bioinfo@sina.com

更新于 2024-09-02
9 篇1 人关注
推荐阅读
公开
3.1_蛋白单体结构预测
songk@dp.tech

发布于 2024-02-29
公开
Protein Alignment
mengyue@dp.tech

发布于 2023-10-12
1 转存文件

