

11: 核磁共振与NMRglue
核磁共振(NMR)光谱学是现代化学中最常见和最强大的分析方法之一。到目前为止,我们主要处理的是基于文本的数据文件,也就是说,可以使用文本编辑器打开并仍包含人类可理解信息的文件。如果你用文本编辑器打开大多数来自 NMR 仪器的文件,它看起来更像是胡言乱语,而不是人类应该能够阅读的东西。这是因为它们是二进制文件;它们是用计算机语言而不是人类语言编写的。
我们需要一个专门的模块来导入和读取这些数据,幸运的是,一个名为 NMRglue 的 Python 模块正好可以做到这一点。该模块包含处理来自各种主要 NMR 光谱文件类型的数据的子模块,包括 Bruker、Pipe、Sparky 和 Varian。它不能读取 JOEL 文件,但现在JOEL光谱仪支持将数据导出为 NMRglue 支持的上述文件类型之一。
目前,Anaconda 不包含 NMRglue,因此您需要自行获取 NMRglue 并安装。以下网站包含安装说明,或者您可以使用 pip 来安装。如果 Jupyter 已经安装在您的计算机上,您应该可以通过终端使用 pip install nmrglue 进行安装,如果您在使用 Google Colab,您应该在笔记本的第一个代码单元格中包含 !pip install nmrglue(请参阅[第 0.2 节])。NMRglue 要求您已经安装了 NumPy 和 SciPy,还应该安装 matplotlib 以进行可视化。
https://nmrglue.readthedocs.io/en/latest/install.html
以下所有使用 NMRglue 的内容都假定使用了包含别名的导入。NMRglue 不是 SciPy 生态系统中的主要库,所以 ng 别名并非严格约定,但在此处使用它是为了方便,也是为了与在线文档保持一致。
11.1 NMR 数据处理
收集 NMR 数据的一般步骤是用射频脉冲激发给定类型的 NMR 活跃核,让它们放松。当它们旋转时,它们的旋转在仪器中引起特征频率的电压振荡,光谱仪记录这些振荡作为下面所示的自由感应衰减(fid)(图 1,左)。我们感兴趣的是这些振荡的频率,因为它们对于受过化学培训的人来说,可以了解核的化学环境。一个挑战是,来自每个核的所有不同信号都堆叠在一起,使得难以区分一个从另一个或确定波频。这类似于计算机在整个乐团同时演奏时辨别单个乐器的问题。幸运的是,有一个名为傅里叶变换的数学方程,它将上述 fid 转换为显示所有不同频率的图形(图 1,右)。这就是所谓的将时域转换为频域。
图 1 原始 NMR 光谱数据通过傅里叶变换从时域(左)转换为频域(右)。
用 Python 处理 NMR 光谱数据的一般步骤如下。
使用 NMRglue 将 fid 数据加载到 NumPy 数组中
将数据傅里叶变换到频域
对光谱进行相位处理
对光谱进行校准
测量峰的化学位移和积分
11.2 使用 NMRglue 导入数据
使用 NMRglue 导入数据是通过表 1 中所示的子模块中的 read 函数完成的。可以在 NMRglue 文档 中找到其他模块。模块的选择取决于数据文件类型。
表 1 NMRglue 模块示例
| 模块 | 描述 |
|---|---|
bruker |
作为单个文件的 Bruker 数据 |
pipe |
作为具有 .fid 扩展名的单个文件的 Pipe 数据 |
sparky |
带有 .ucsf 扩展名的 Sparky NMR 文件格式 |
varian |
作为具有 .fid 扩展名的数据文件夹的 Varian/Agilent 数据 |
jcampdx |
具有 .dx 或 .jdx 扩展名的 JCAMP-DX 文件 |
read() 函数加载 NMR 文件并返回一个包含元数据字典和 ndarray 中数据的元组。字典包括完成 NMR 日期处理所需的信息。查看下面的 NMR 数据,您可能注意到每个点都包括实部和虚部(即带有 j 的数学术语)。稍后在对光谱进行相位处理时,两者都是必需的,所以不要丢弃任何数据。
array([-0.00194889-0.00471539j, -0.00192186-0.00472489j,
-0.00191337-0.00473085j, ..., -0.00189737+0.00591656j,
-0.00191882+0.005872j , -0.00191135+0.00587132j], dtype=complex64)本演示中使用的数据已经在光谱仪上进行了傅里叶变换,因此以下单元格为演示目的而反转此过程。有些光谱仪会自动进行傅里叶变换,而有些则不会。
上面的字典 dic 包含一个非常长的值列表,不同文件格式之间的字典键可能会有所不同。为了保持一个更短、更有用且更一致的元数据字典,NMRglue 提供了 guess_udic() 函数,用于在所有文件格式之间生成一个通用字典。
{'ndim': 1,
0: {'sw': 5994.65478515625,
'complex': True,
'obs': 399.7821960449219,
'car': 1998.9109802246094,
'size': 13107,
'label': 'Proton',
'encoding': 'direct',
'time': False,
'freq': True}}通用字典是一个嵌套字典。第一个键是 ndim,它提供了 NMR 光谱中的维度数量。大多数 NMR 光谱是一维的,但二维的也相当普遍。随后的键是 NMR 光谱中的每个维度,值为嵌套的元数据字典。因为上面的光谱数据是一维的,所以只有一个嵌套字典。下表 2 提供了通用字典中包含的每个元数据的描述。
表 2 单维度 udic 字典键*
| 键 | 描述 | 数据类型 |
|---|---|---|
car |
载波频率 (Hz) | 浮点数 |
complex |
指示数据是否包含复数值 | 布尔值 |
encoding |
编码格式 | 字符串 |
freq |
指示数据是否在频率域中 | 布尔值 |
label |
观察到的核 | 字符串 |
obs |
观察频率 (MHz) | 浮点数 |
size |
光谱中的数据点数 | 整数 |
sw |
频谱宽度 (Hz) | 浮点数 |
time |
指示数据是否在时域中** | 布尔值 |
* 也就是说,我们假设我们正在查看来自 NMR 数据的单个维度,所以例如,我们正在查看 udic[0]。
** 鉴于数据必须位于频率或时域中,freq 和 time 关键字实际上提供了相同的信息。
11.3 傅里叶变换数据
当数据首次导入时,它通常位于时域中。您可以通过检查 udic 中的 time 值是否设置为 True 来确认这一点,如下所示。
udic[0]['time']
我们还可以通过使用 matplotlib 绘制来查看数据。
要将数据转换为频率域,我们将使用来自 fft SciPy 模块的快速傅里叶变换函数(fft)。NMRglue 也包含傅里叶变换函数,但我们将在这里使用 SciPy。下面的图形通过以下方式反转 x 轴:
当你绘制傅里叶变换后的数据时,您可能会收到一个 ComplexWarning 错误消息,因为傅里叶变换将返回复数值(即具有实部和虚部的值)。要仅使用实部分量,请使用上面所做的 .real 方法。现在,图像看起来更像 NMR 光谱,但大部分共振是失去相位的。下一步是对光谱进行相位校正。
11.4 相位校正数据
相位校正是使所有峰值向上指向的后处理过程,如图 2 所示。这不仅仅是取绝对值,因为这样并不总是能产生单一峰值,所以 NMRglue 包含了一系列用于相位校正光谱的函数。
图 2 对 NMR 光谱进行相位校正会使所有信号指向正方向。
11.4.1 自动相位校正
相位校正 NMR 光谱的最简单方法是让自动相位校正函数为您处理。下面的函数接受数据和相位校正算法作为参数。
ng.process.proc_autophase.autops(data, algorithm)
允许的相位校正算法可以是 acme 或 peak_minima。向 autops() 函数提供带有实部分量和虚部分量的数据数组非常重要。
Optimization terminated successfully.
Current function value: 0.001729
Iterations: 120
Function evaluations: 238
你应该尝试这两种算法,看看哪种对你最有效。上面的光谱是使用 acme 自动相位校正算法得到的,结果接近但仍然有些偏差。如果这两种自动相位校正算法都不适用于你,那么你需要按照下面的讨论手动相位校正 NMR 光谱。
11.4.2 手动相位校正
手动相位校正 NMR 光谱分为两个步骤。首先,需要调用 manual_ps phasing() 函数并调整 p0 和 p1 滑块,直到光谱看起来具有相位校正。
%matplotlib # exists inline plotting
p0, p1 = ng.process.proc_autophase.manual_ps(fdata.real)
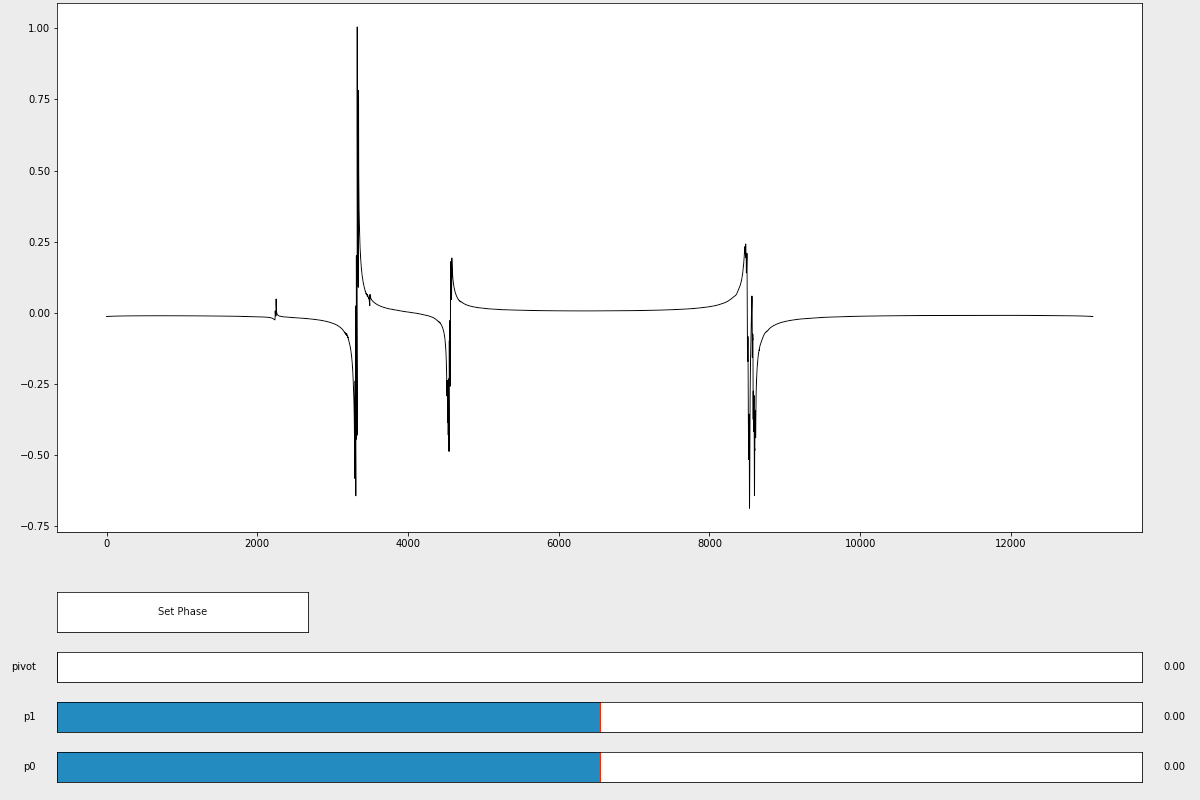
关闭窗口后,函数将返回 p0 和 p1 的值,您发现这些值可以正确地相位校正光谱。第二步,将这些 p0 和 p1 值输入到 ps() 相位校正函数中,以实际相位校正光谱。
phased_data = ng.proc_base.ps(fdata, p0=p0, p1=p1)
%matplotlib inline # reinstates inline plotting
fig3 = plt.figure(figsize=(16,6))
ax3 = fig3.add_subplot(1,1,1)
ax3.plot(phased_data.real)
plt.gca().invert_xaxis()
然后,你可以绘制 phased_data 以获得具有所有峰值向上指向的 NMR 光谱。
11.5 化学位移
尽管 NMR 光谱现在已经相位校正,但它不太可能被正确地参照。也就是说,峰值目前并未位于正确的化学位移上。参照通常是通过了解溶剂共振或内部标准(如四甲基硅烷,TMS)的公认化学位移,并通过校正因子调整光谱来进行的。目前,我们是根据每个数据点的索引绘制数据,所以首先我们需要创建一个频率缩放的x轴数组,然后调整光谱的位置,使其正确参照。
11.5.1 生成 X 轴
x轴是频率刻度,因此这个轴有时以赫兹(Hz)表示。然而,由于 NMR 共振频率取决于仪器场强,因此相同的样品在不同的仪器中会产生不同的频率。为了使频率轴独立于谱仪场强,NMR 光谱通常以ppm 比例呈现,即观测到的化学位移(Hz)与观测到的特定核的谱仪频率(MHz)之间的比值。
这使得从谱仪到谱仪的峰值位置始终保持一致,无论磁场强度如何。这就是第 11.2 节中 udic 的重要性所在,因为我们可以获得光谱的观测频率宽度(Hz)和数据的分辨率。后者是光谱中有多少个数据点,这一点很重要,以便我们避免绘图错误(我们都知道这个错误:ValueError: x and y must have same first dimension, ...)。如果 udic 中的任何值为 999.99,这意味着谱仪没有记录这部分信息,您需要在其他地方找到它。
现在,如果我们绘制光谱,我们会看到它是以ppm刻度显示的。
另外,NMRglue包含了一个称为单位转换对象的对象,可以用于在NMR光谱中的任何位置之间转换ppm、Hz和点索引值。要创建一个单位转换对象,请使用make_uc()函数,该函数需要两个参数 - 从第11.2节中读取的NMR文件生成的字典dic和原始数据数组data。
如果您使用的是与pipe不同的NMR文件格式,请将`pipe`更改为表1中的适当格式。
unit_conv = ng.pipe.make_uc(dic, data)
ppm = unit_conv.ppm_scale()
上述代码的最后一行生成了用于绘制 NMR 数据的 x 轴所需的 ppm 值数组。以下示例使用我们自己计算出的 ppm 刻度。
11.5.2 参照数据
在上面的光谱中,2.58 ppm 处的小共振是内部 TMS(四甲基硅烷)标准,应位于 0.00 ppm。诱惑是从 x 轴减去 2.58 ppm,但光谱不仅仅是移动,而是滚动。也就是说,当光谱移动时,其中一部分会从一端消失并在另一端重新出现(图 3)。
图 3 参照 NMR 光谱是通过滚动它,直到峰值位于正确的位移位置。当信号从光谱的一端掉落时,它会在另一端重新出现。
对我们来说方便的是,NumPy 有一个 np.roll() 函数,它可以将数据精确地滚动到数组数据中。
np.roll(array, shift)
np.roll() 函数需要两个参数。第一个是包含数据的数组,第二个是移动或滚动数据的量。位移不是以 ppm 为单位,而是以数据数组中的位置为单位。如果您知道以 ppm 为单位的参照校正(Δppm),请使用以下方程,该方程描述了 ppm 校正(Δppm)和数据点数量校正(Δpoints)之间的关系。size 是光谱中的点数,obs 是观测到的载波频率,sw 是扫描宽度(以 Hz 为单位)。这些值都可以从通用字典中获得。
下面的示例需要将光谱移动 -2.58 ppm。
如果您想将绘图范围缩小到共振所在的位置,可以使用 plt.xlim(8,0) 函数。注意,首先是8,表示绘图范围从 8 ppm 0 ppm。使用 plt.xlim(8,0) 可以避免使用 plt.gca().invert_xaxis() 来翻转 x 轴。
11.6 积分
可以使用 scipy.integrate 模块中的积分函数或 NMRglue 的积分函数来对峰下面积进行积分。由于 NMRglue 中的积分函数支持 ppm 比例中的极限值,所以它可能是最方便的,如下所示。
使用下面的 integrate() 函数执行积分,其中 data 是作为 NumPy 数组的 NMR 数据,conv_obj 是 NMRglue 单位转换对象(参见第 11.5.1 节),而 limits 是积分极限的列表或数组。
ng.analysis.integration.integrate(data, conv_obj, limits)
极限值以 ppm 为单位,因此观察上面的光谱并决定您想要放置积分极限的位置。上面的 NMR 光谱显示了选定的积分极限,这些极限以红色垂直线表示。
现在开始对我们的 NMR 光谱进行积分。
array([-0.00061348, -0.00035478, -0.00034138], dtype=float32)
这些值可能不是你预期的,但是如果我们将所有值除以最小值,我们就会得到峰下相对面积,就像你从大多数 NMR 处理软件中获得的那样。
array([1. , 0.5783051 , 0.55646706], dtype=float32)
上图是在中的乙基苯的 NMR光谱,其中有五个芳香质子,另外两个共振应该有三个和两个质子。如果我们进行一些数学计算,使积分总和为十个质子,并四舍五入到最接近的整数,我们得到 5:3:2。可能由于溶剂共振(,7.27 ppm)被包含在芳香质子的积分中,误差较小。
11.7 峰值拾取
从 NMR 光谱中提取的另一条常见信息是共振的化学位移。与积分类似,SciPy 包含了诸如 scipy.signal.argrelmax() 之类的函数,可以在光谱中找到峰值,但同样,NMRglue 包含了一个专门用于在 NMR 光谱中定位峰值的函数,如下所示。
ng.analysis.peakpick.pick(data, pthres=)
峰值拾取函数有许多可选参数,但是必须提供两个必要的信息:data 数组和一个正阈值(pthres),超过该阈值的任何峰值都将被识别。观察下面的光谱,所有峰值都在0.1(绿色虚线)以上,基线在0.1以下,因此这看起来是一个合理的阈值。
`ng.analysis.peakpick.pick()` 目前无法与 NumPy 1.24 及更高版本一起使用。如果您需要使用此功能,请将 NumPy 降级到 1.23 或更早版本。
rec.array([(1077., 1, 34.1857841 , 27.63016701),
(2299., 2, 31.36229494, 16.87085152),
(2332., 3, 5. , 0.88378525),
(6275., 4, 35.96975553, 25.60304451),
(6355., 5, 32.1389443 , 17.82140732)],
dtype=[('X_AXIS', '<f8'), ('cID', '<i8'), ('X_LW', '<f8'), ('VOL', '<f8')])此函数的输出是一个元组数组,每个元组包含有关识别的峰值的信息。从这里我们已经可以看出有四个峰值被识别。每个元组包含峰值的索引、峰值编号、峰值的线宽以及每个峰值面积的估计。我们可以使用索引值来索引 ppm 数组以获取化学位移。
[1.2320797763091693, 2.6300384454361936, 2.6677901934568085, 7.178552085738197, 7.2700714748790825]
我们可以绘制带有这些化学位移的 NMR 光谱,这些化学位移用下面的垂直虚线标记。看起来它在定位共振方面做得相当好!如果 NMRglue 未能正确识别峰值,可以调整 NMRglue文档 中描述的许多参数。
延伸阅读
NMRglue 官网:https://www.nmrglue.com/(免费资源)
NMRglue 文档页面:http://nmrglue.readthedocs.io/en/latest/tutorial.html(免费资源)
J.J. Helmus, C.P. Jaroniec, Nmrglue: An open source Python package for the analysis of multidimensional NMR data, J. Biomol. NMR, 2013, 55, 355-367, http://dx.doi.org/10.1007/s10858-013-9718-x.(关于 nmrglue 的论文)
练习
使用 NMRglue 打开在 中使用 TMS 测量的乙醇(EtOH_1H_NMR.fid)的 1H NMR 光谱。使用
pipe模块。a) 绘制结果光谱,并确保正确引用(如果尚未完成)。
b) 积分甲基()与亚甲基(--)共振,并计算比值。
- 打开 2-乙基-1-己醇的 和 NMR 光谱,2-ethyl-1-hexanol_1H_NMR_CDCl3.fid 和 2-ethyl-1-hexanol_13C_NMR_CDCl3.fid,它们是在 中使用 TMS 测量的。在 ppm 刻度上绘制它们。如果尚未完成,请确保正确调整相位并引用光谱。使用
pipe模块。










