

Uni-MM PB/GBSA Structure
导言
分子力学泊松-玻尔兹曼表面积(MM/PBSA)和分子力学广义玻恩表面积(MM/GBSA)是近10年应用最为广泛的结合自由能预测方法之一。相比大多数分子对接的评分函数,MM GB/PBSA更精确,计算量更少,是目前结合自由能预测最流行的方法。
Uni-MM PB/GBSA模块能够比较准确预测化合物抑制剂的结合自由能,以及不同化合物抑制剂的结合自由能排序,进而理解不同化合物抑制剂和蛋白结合的效果。
在本教程中,你将学习使用Uni-MM PB/GBSA模块预测SARS-CoV-2主要蛋白酶(SARS-CoV-2 Main Protease)与5种虚拟筛选的化合物抑制剂的结合自由能 [1],理解化合物抑制剂对SARS-CoV-2 Main Protease的结合亲和力。
此教程中使用的蛋白结构文件:
此教程中使用的化合物结构文件:
7O46_17.sdf
本教程中使用的化合物抑制剂的结合能实验值:
| PDB ID | Compound | IC50(μM) |
| 7O46 | 17 | 0.15 |
| 7NEO | 15 | 6.60 |
| 7B2J | 5 | 7.20 |
| 7B5Z | 6 | 38.50 |
| 7B77 | 8 | 79.30 |
1. 导入结构
- 左侧通用菜单栏 Menu → File→ Import Structure
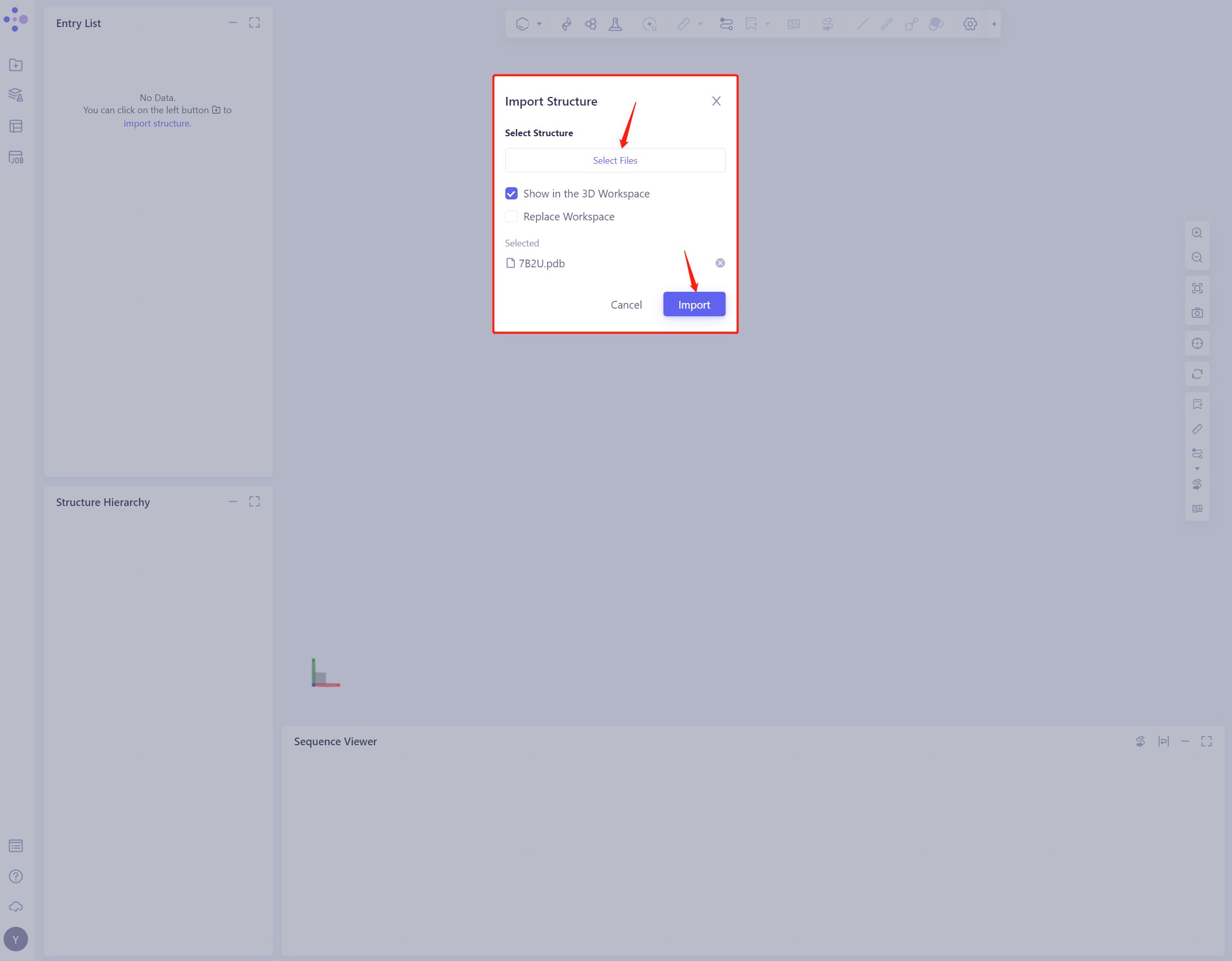
- 点击Select Files,选择7B2U.pdb文件,点击Import导入该蛋白结构
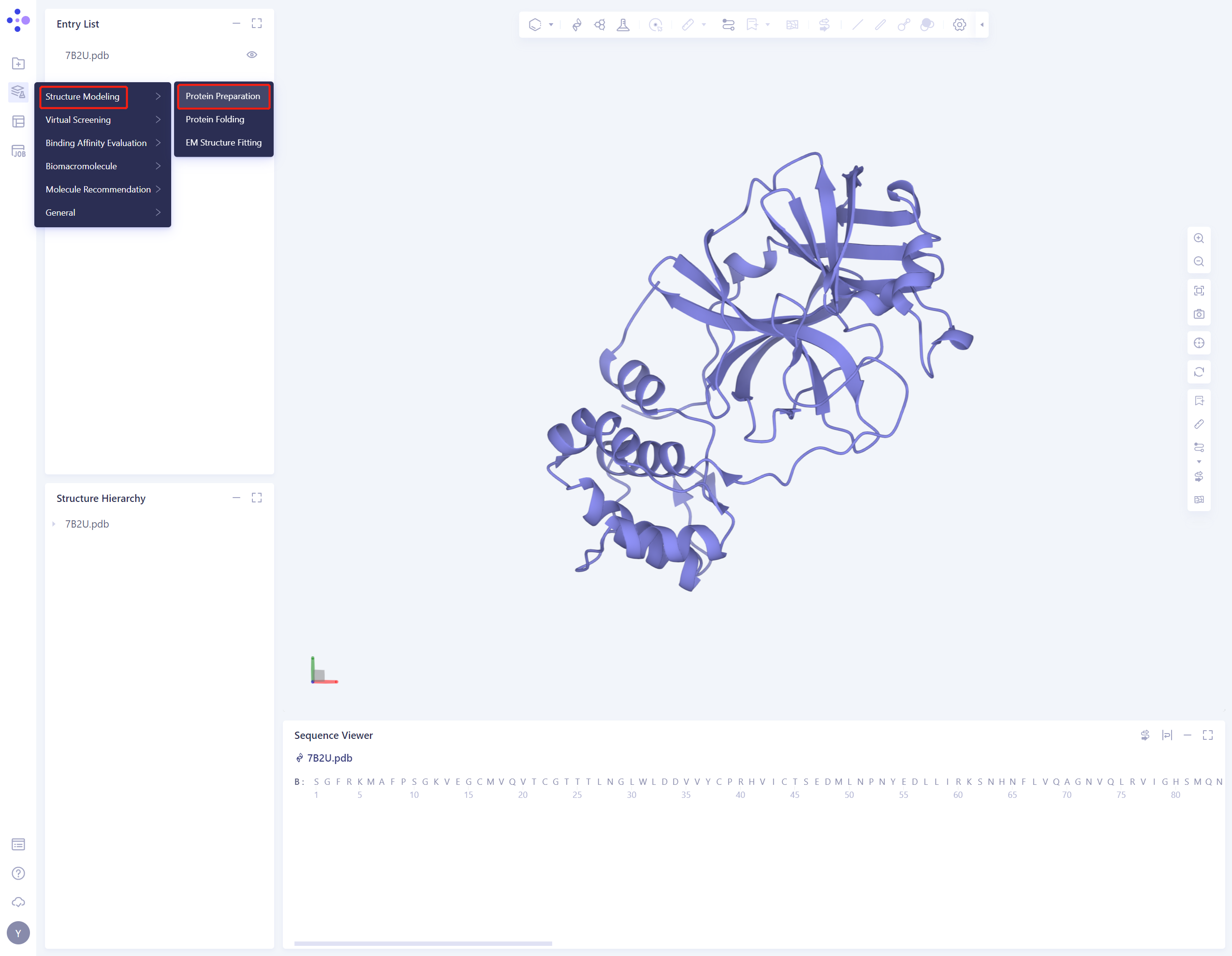
2. 体系准备
2.1 准备蛋白结构
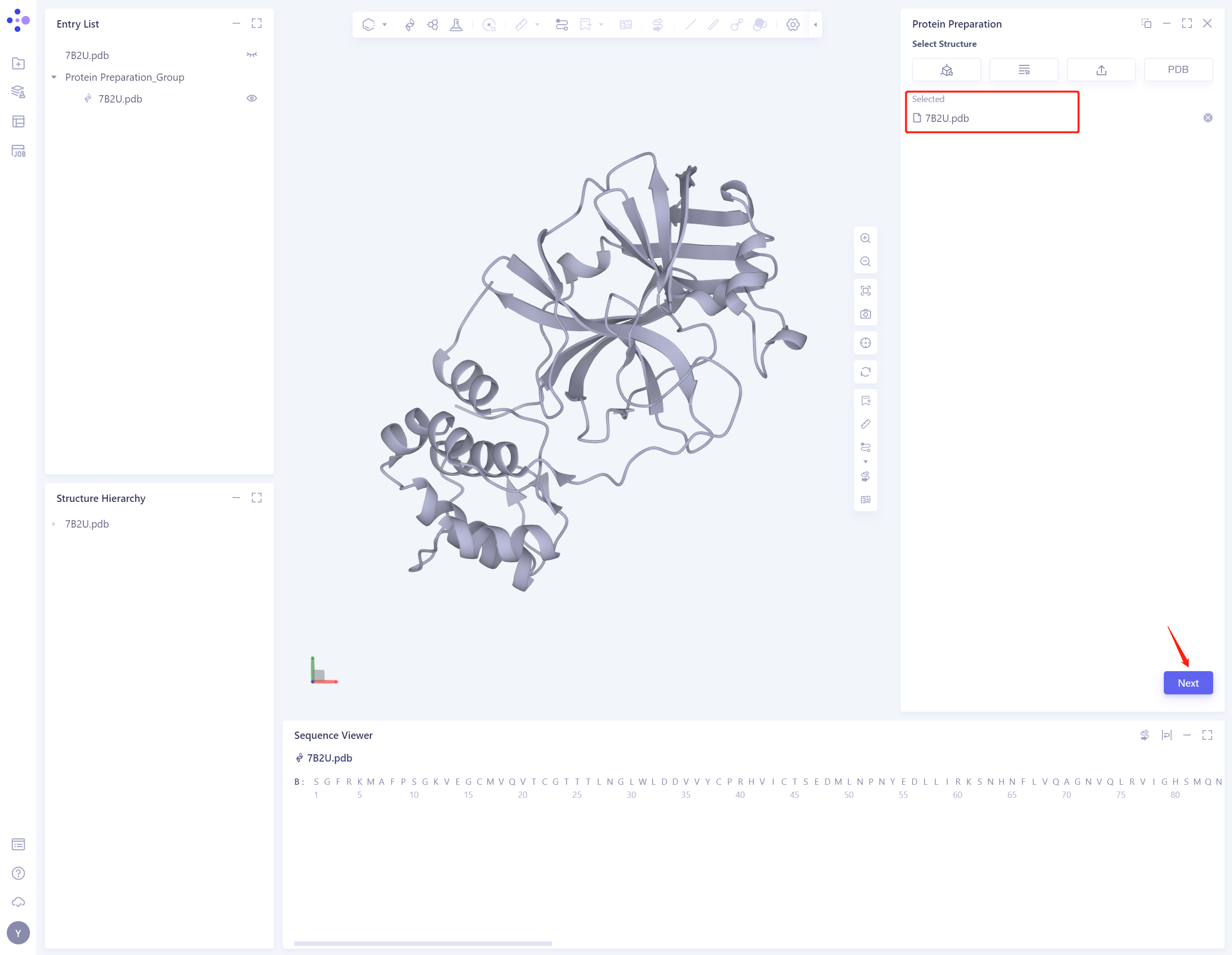
2.1.1 Select Structure
- 左侧通用菜单栏Function → Structure Modeling → Protein Preparation
- Select Structure from 3D Workspace:在Structure Hierarchy中选中Protein,点击Ok
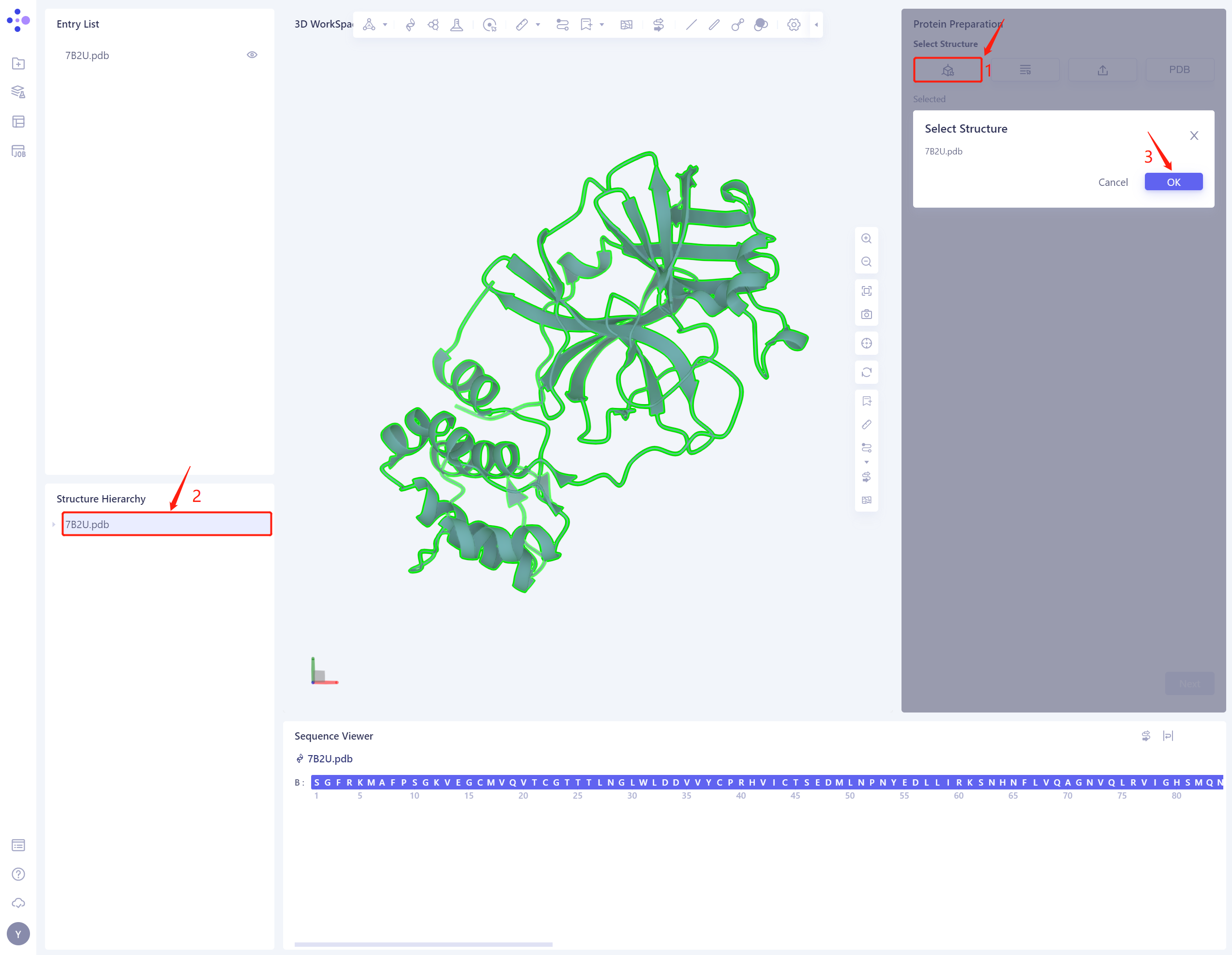
- 7B2U.pdb被加载到Protein Preparation参数设置面板,点击Next
2.1.2 Select Polymer、Other Groups to Keep
- 选中B链蛋白,并点击 Next(此蛋白结构中无other groups)
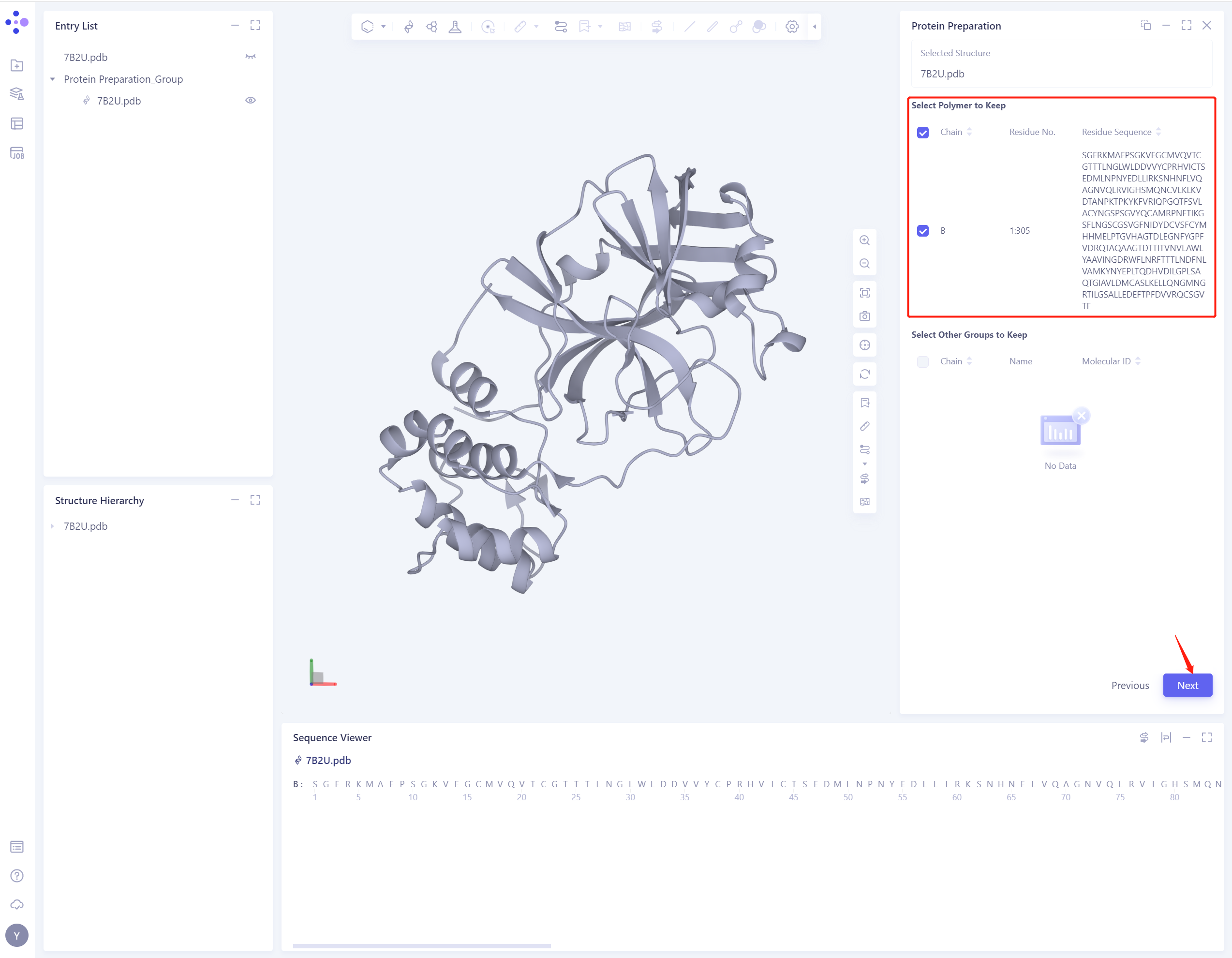
2.1.3 Select Missing Residues to Repair
- 本案例中无Missing Residues
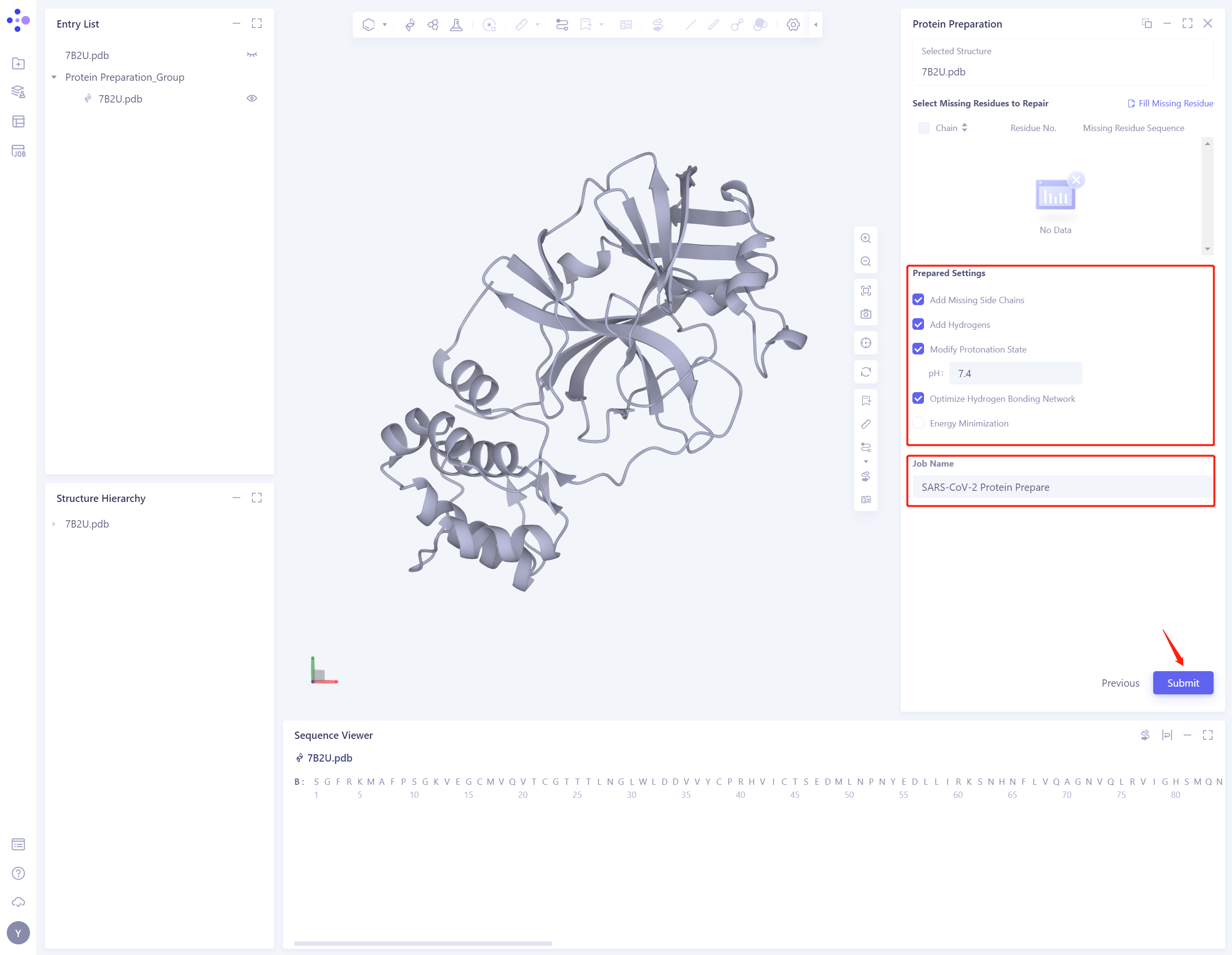
2.1.4 Prepared Settings
- 设置参数详见 2.2.5中的图,取消勾选Energy Minimization选项
2.1.5 命名Job并提交任务
- Job Name命名为“SARS-CoV-2 Protein Prepare”,点击“Submmit”提交任务
2.1.6 查看蛋白准备结果
- Protein Preparation计算任务一般在十几秒到几分钟内完成。任务完成后,通过点击Jobs,查看相应任务;
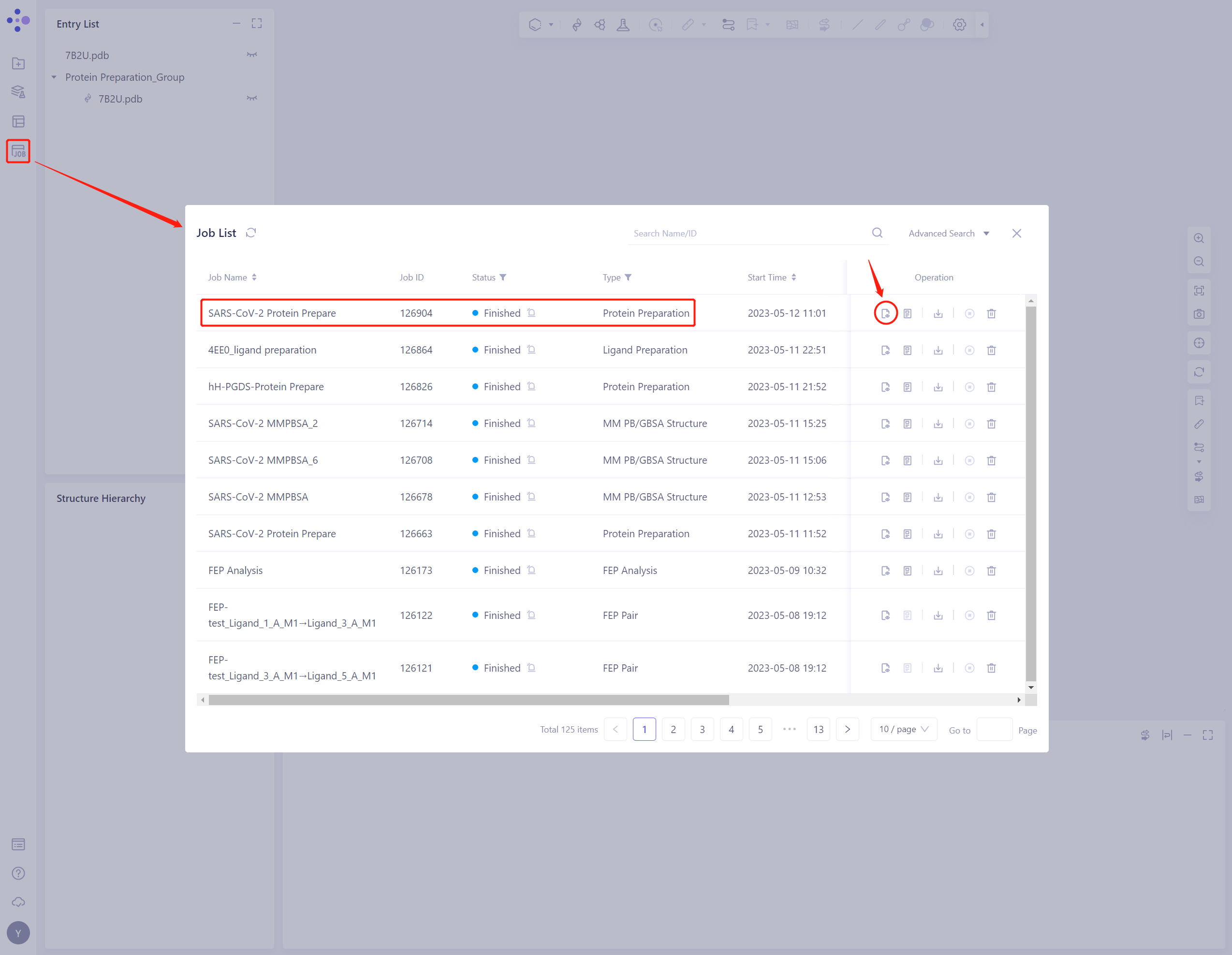
- 点击“show”按钮,将准备后的蛋白结构展示在3D Workspace内
3. 创建MM PB/GBSA structure任务
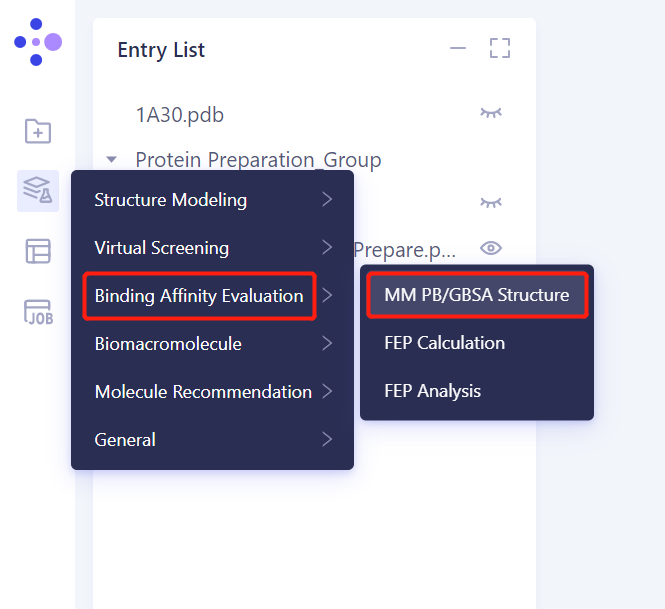
3.1 入口
左侧通用菜单栏 Menu → Function → Binding Affinity Evolution → MM PB/GBSA Structure
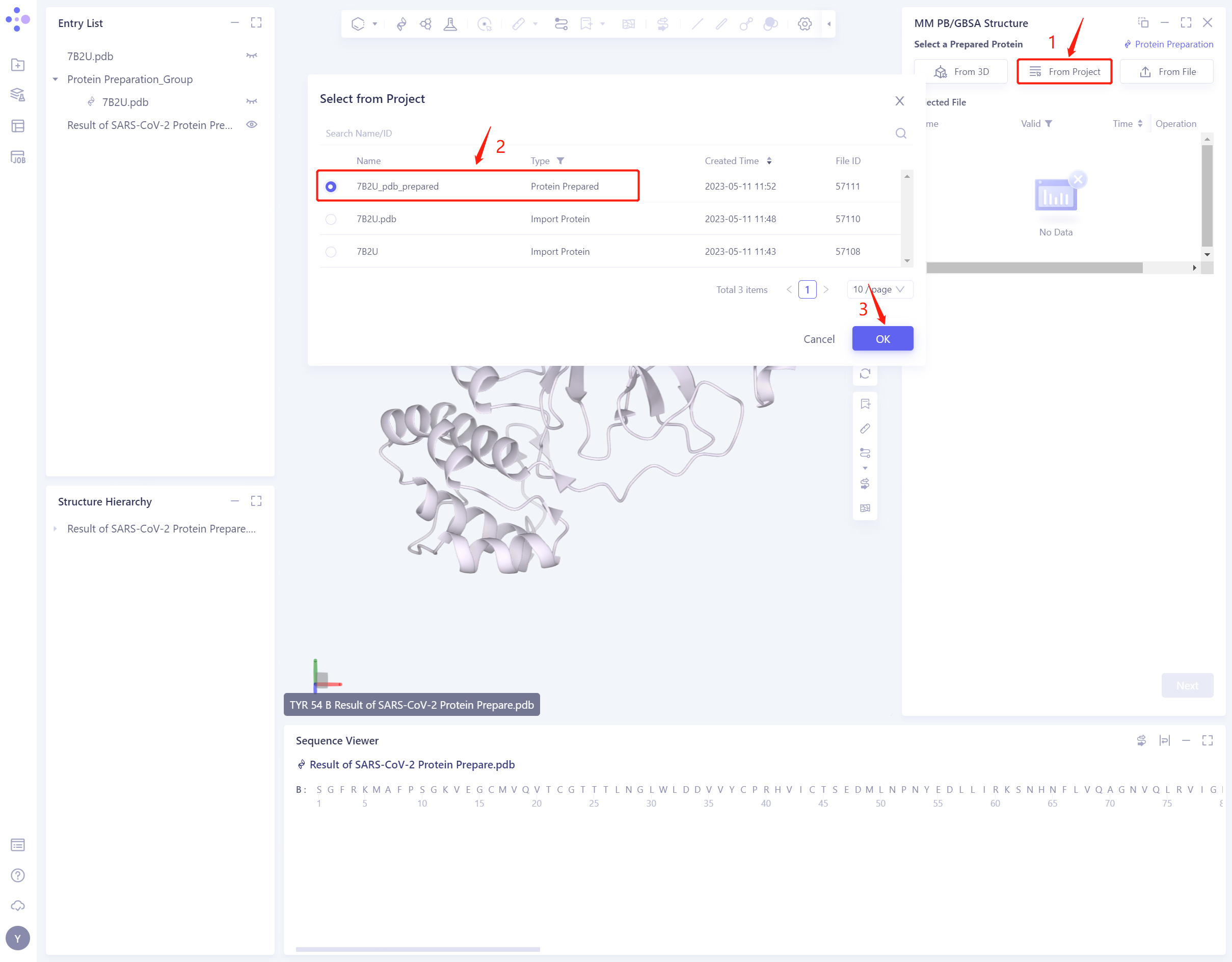
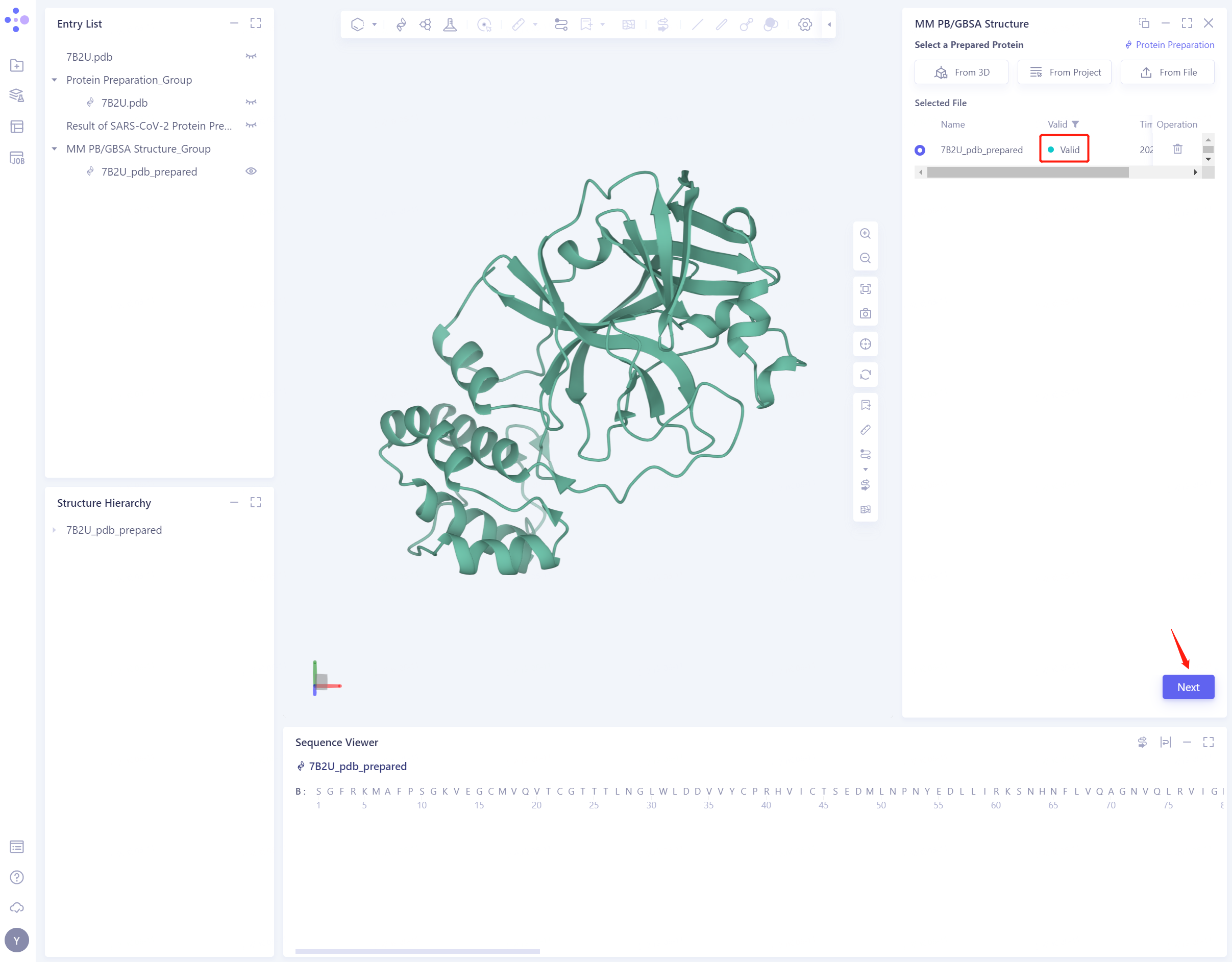
3.2 Select Prepared Protein From Project
- 选择 “7B2U_pdb_prepared” 作为准备后的蛋白结构,点击 OK
- 点击 OK 后,系统会自动检查输入的蛋白是否符合计算要求,状态为“Processing”
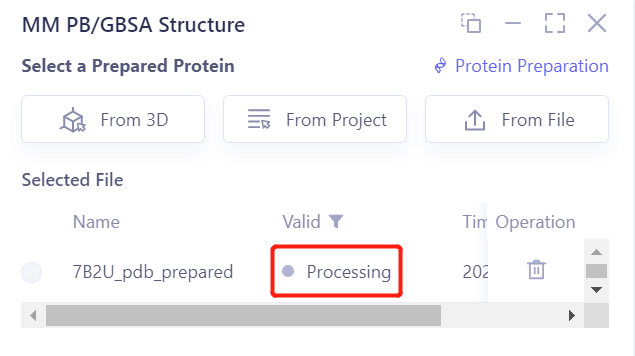
- 大约不到1分钟,系统会判断该蛋白为“Valid”状态,点击“Next”
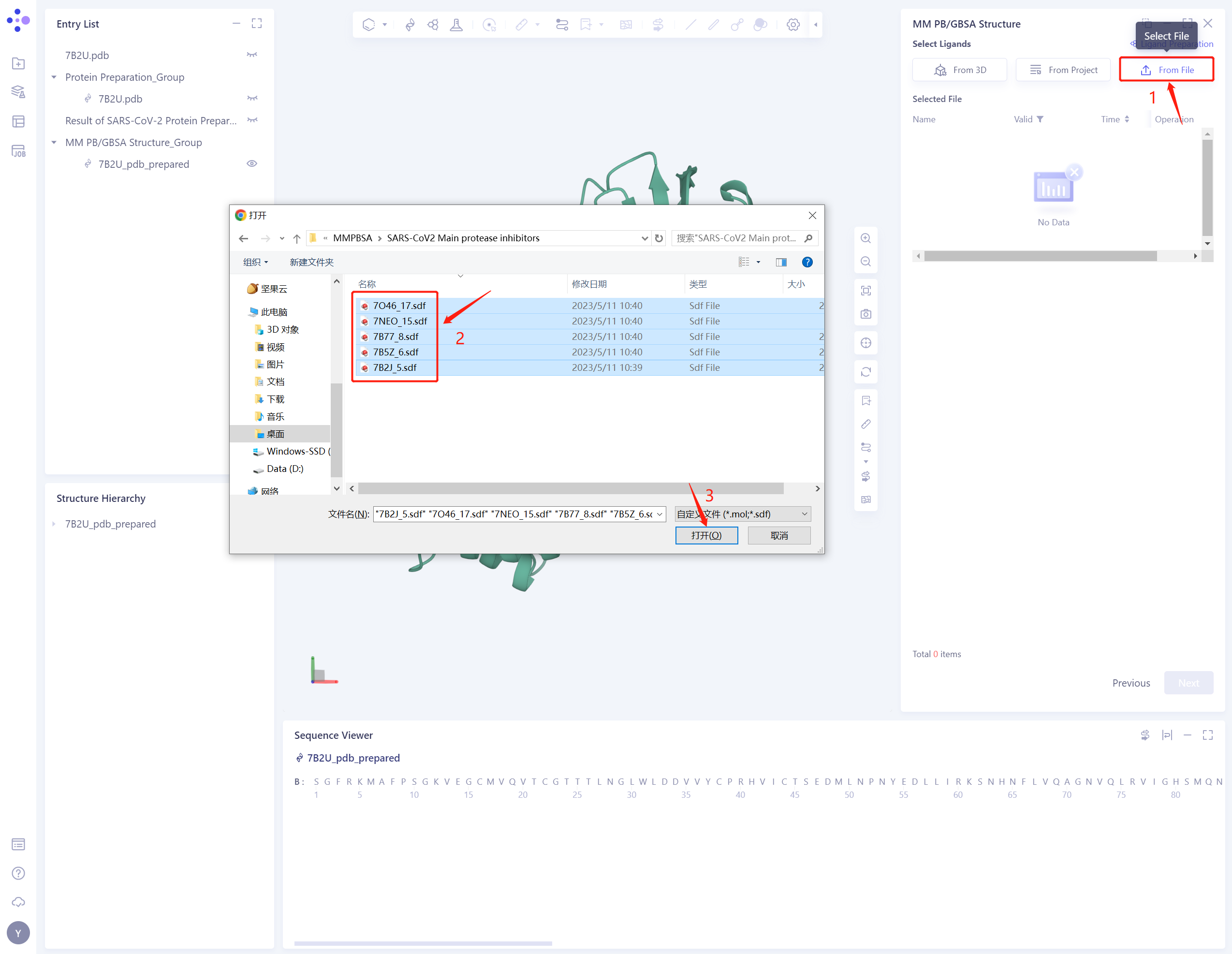
3.3 Select Ligands
点击 “From File”;
在本地文件夹中选中7O46_17.sdf、7NEO_15.sdf、7B77_8.sdf、7B5Z_6.sdf、7B2U_1.sdf、7B2J_5.sdf化合物结构;
点击“打开”,导入配体结构。
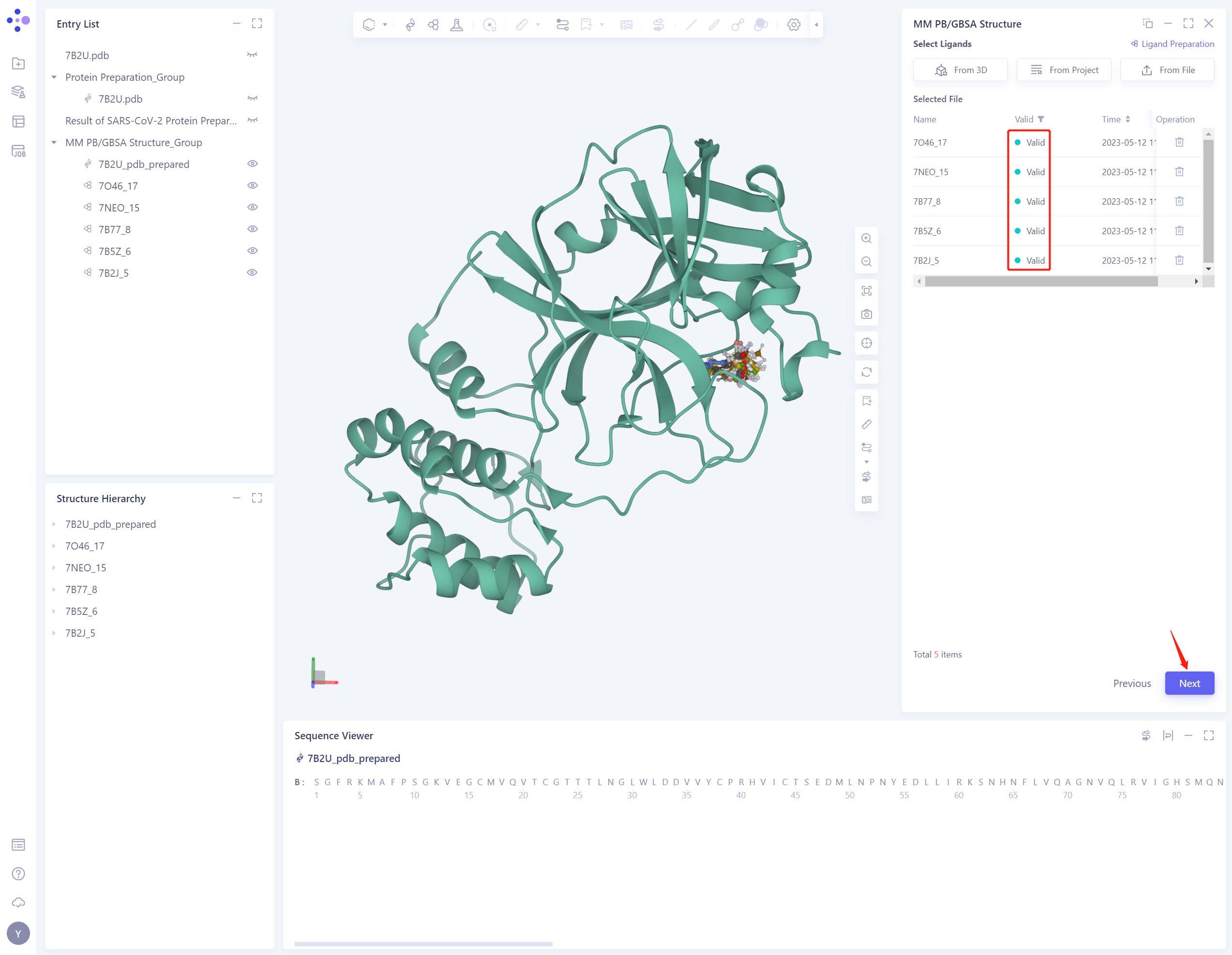
5个配体结构将被载入 “Selected File”。系统会自动检查输入的Ligand是否符合计算要求,状态为“Processing”,随后,系统会判断该配体为“Valid”状态。
点击Next
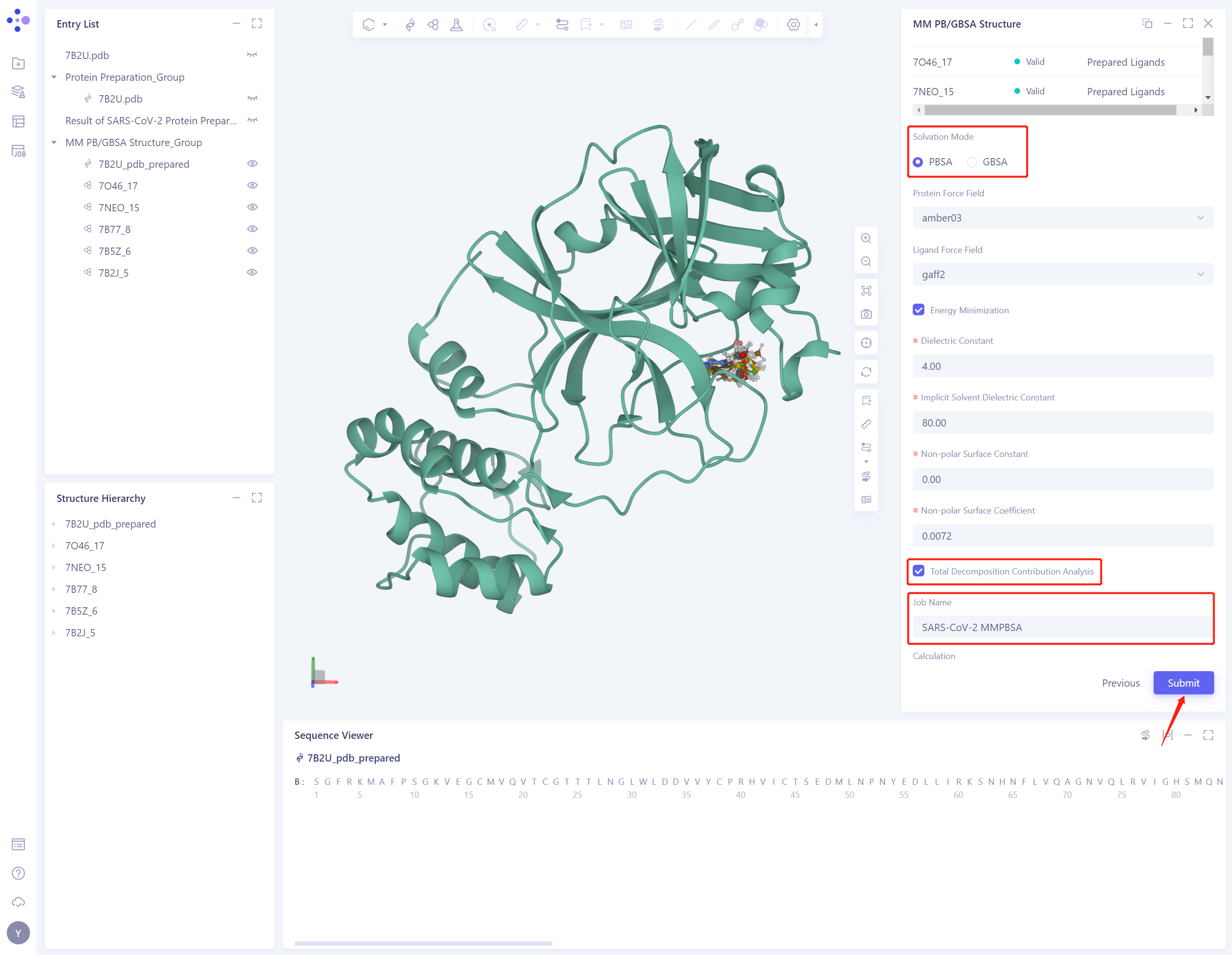
3.4 Confirm and Setting
本教程中,Solvation Mode选择“PBSA”;
勾选Total Decomposition Contribution Analysis选项,对残基能量进行分解;
“Job Name” 命名为 “SARS-CoV-2 MMPBSA”;
其余参数保持默认值,点击“Submmit”提交任务。
4. 结果分析
4.1 入口
- 左侧通用菜单栏Job → Job List,找到“SARS-CoV-2 MMPBSA”计算任务,点击 “Show”
4.2 结果展示
计算得到的结合自由能结果顺序如下图所示。通过和实验IC50值对比,5种化合物抑制剂的结合能预测排序均比较准确。
结果表明,Uni-MM PB/GBSA模块能够比较准确预测化合物抑制剂的结合自由能,进一步可以用于预测化合物抑制剂和蛋白的结合亲和力,为药物设计和优化过程提供理论支持。
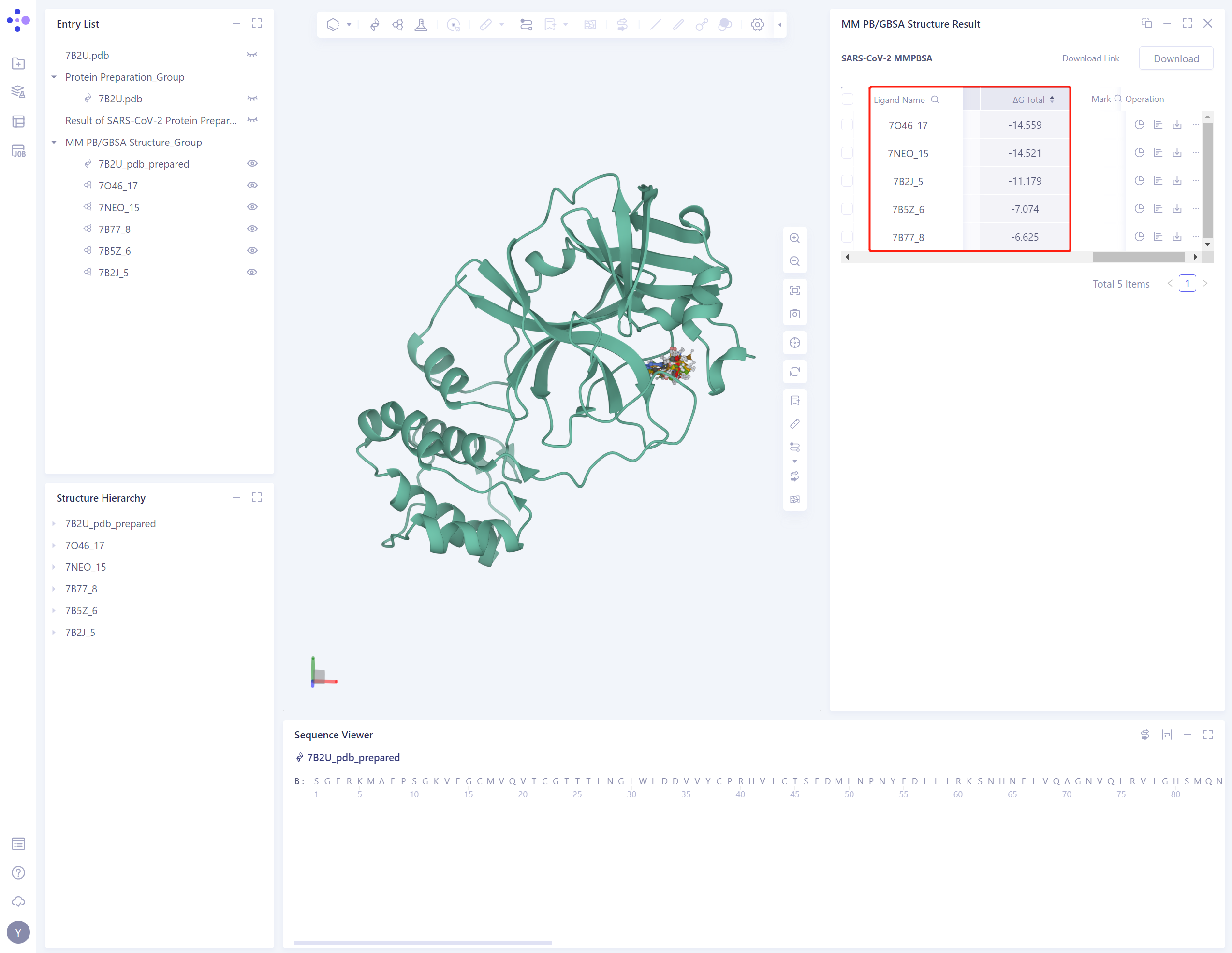
- 本教程中使用的化合物抑制剂的结合能实验值:
| PDB ID | Compound | IC50(μM) | MM PBSA (kcal/mol) |
| 7O46 | 17 | 0.15 | -14.559 |
| 7NEO | 15 | 6.60 | -14.521 |
| 7B2J | 5 | 7.20 | -11.179 |
| 7B5Z | 6 | 38.50 | -7.074 |
| 7B77 | 8 | 79.30 | -6.625 |
5. 参考文献
[1] Ultralarge Virtual Screening Identifies SARS-CoV-2 Main Protease Inhibitors with Broad-Spectrum Activity against Coronaviruses. J. Am. Chem. Soc. 2022, 144, 7, 2905–2920.





