

作者:Changxin Wang
时间:2023.11.8
推荐镜像:abacus-user-guide:3.3.2
推荐计算资源:CPU。
内容:本教程力图深入浅出的介绍材料微观到宏观的基本概念,建立微观属性和宏观属性的联系。
使用方式:您可在 Bohrium Notebook 上直接运行。您可以点击界面上方蓝色按钮 开始连接,选择 abacus-user-guide:3.3.2 镜像及2核心及以上节点配置,稍等片刻即可运行。 如您遇到任何问题,请联系 bohrium@dp.tech。
共享协议:本作品采用知识共享署名-非商业性使用-相同方式共享 4.0 国际许可协议进行许可。
导读
微观到宏观,我们需要跨越什么。这可能是一个非常普遍的问题,普遍到可能每个人都能说出自己的观点。什么缺陷,扩散,位错、晶界、相变、析出、偏析、缺陷、织构、畴界、内应力、blablabla。
但是至少我们能达到一条共识,宏观绝对不是微观世界的叠加。由于尺寸效应存在,宏观性能和微观性能往往差距很大,两者之间的尺寸跨越称为介观尺度。 在介观尺度,我们方面可以看到由于微观晶体结构差异导致的相形成差异,也能从介观的晶粒信息估计断裂倾向等宏观力学性能。所以教科书上说的,电学性能决定在微观(不绝对),力学性能决定在介观,也充分证明了介观尺度的重要性。
Oh,那我们跨越介观不就行了? 然而,介观位于纳米(10-9米)到微米(10-6米)之间,尺度几乎跨越了几百个原子并排长度到可以放大镜观察的头发直径尺寸。我们都知道,微观世界可以用量子力学、密度泛函、分子动力学模拟,重点关注完美的晶体、原子、键信息,突出一个完美(别杠,杠就给你搞掺杂,<_<)。宏观世界可以用有限元模拟,突出一个建模均匀(还杠,那就加点梯度变化,加个预制裂纹)。这也就是说,所有的突变、不完美的、模糊不清的机制都被放到了介观尺度下。所以我们怎么跨越介观尺度是个值得考虑,但会掉头发的问题。
1.本文主要针对于固体凝聚态,其他的也不懂啊。
2.微观介观宏观具体尺寸划分没那么清楚,相互扯皮。
3.wangchagnxin boliqq纯手打,转载请注明出处。
完美晶体

首先,我们先以一个简单的材料——单晶硅,直观地理解微观到介观的跨越过程。上图所示是一个完美晶体的单晶硅电镜照片,它属于立方晶系,金刚石结构,空间群为Fd3m。
如果不出意外的话,它是99.9999%以上纯度的硅原子堆叠而成,从1700多度液态硅中拉拔出来(https://baike.so.com/doc/6560022-6773776.html) 。经过一系列后续工艺,出现在你的手机里。
现在在微观层面,搭建下金刚石结构的输入文件,算下它的能量、原子键长、弹性矩阵、层错能等,相信对大家非常容易。
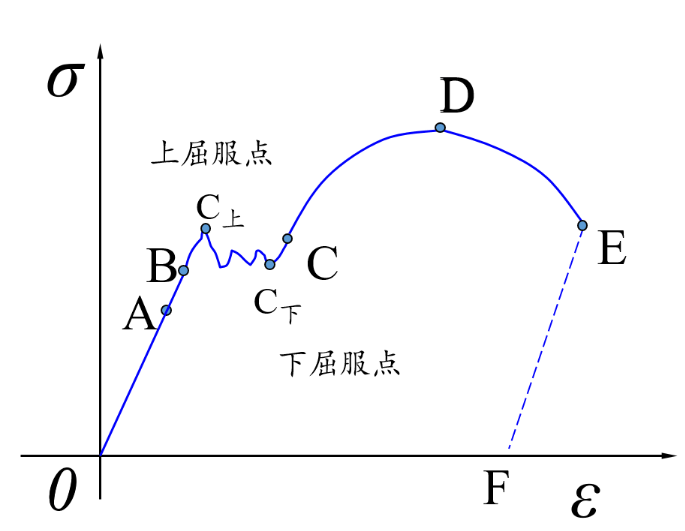
而介观上。。。哇,好像什么也不用干,他太完美了!这导致宏观的性质基本可以从微观性质对应得到。比如宏观的弹性参数矩阵矩阵可以直接对应到微观的晶格弹性矩阵。如下图所示,整个晶体的应力应变曲线基本为一条直线,没有屈服现象和塑性变形,超出弹性极限会直接拉断。材料发生断裂时候的强度与原子键强相关。

位错
**家人们,上强度!**我们先从晶体内部加点料吧,是吧,位错。
位错指晶体材料的一种内部微观缺陷,即原子的局部不规则排列(晶体学缺陷),通常外在的表现是原子层与层之间有线状的对不齐。
在没有位错存在的区域,电子通过等间距规则排列的各晶面时将可能发生衍射,其衍射角、晶面间距及电子波长之间满足布拉格定律(Bragg’s law)。 由于位错存在,原先完美的统一的原子间距不再统一,位错存在的区域附近,晶格发生了畸变,因此衍射强度亦将随之变化,于是位错附近区域所成的像便会与周围区域形成衬度反差,用透射电子显微镜下观察位错位错附近区域所成的像,便会与周围区域形成衬度反差,图象上便出现了一条条的线。位错可长可短,短的位错几十个原子尺度,长的位错在高分辨显微镜下可以直接观察到。如下如所示。
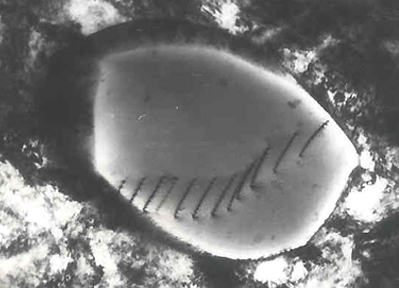
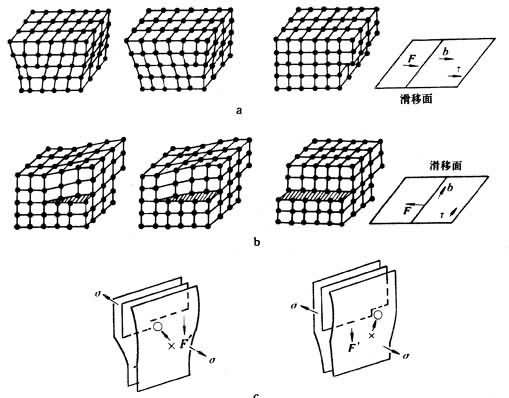
位错具有多种类型,刃型位错、螺型位错、混合位错等。通常起源于内应力或者外部塑性变形导致的原子错配等多种机制。反过来,这些产生的位错将导致原子之间的结合力发生变化,即位错又会导致材料的形变能力发生改变。如施加塑性变形应力增加位错,释放后,再次施加应力,发现弹性常数增加,这便是称为加工硬化现象。这里的内应力产生原因如,在晶体形核长大过程中,由于成分偏析导致的原子间距尺寸的变化过大,或者温度导致的尺寸收缩等等。
位错也导致了 屈服现象 出现,材料内部存在的大量位错,在刃型位错线附近原子发生畸变,从而使能量升高,晶体处于不稳定状态。溶质原子会“自觉”聚集到刃位错受拉部分,因为这样会降低晶格的畸变,使体系更加稳定。此时,位错原子如果想要运动就要克服更大的阻力,这种现象就是所谓的柯氏气团。柯氏气团的出现会影响位错在外力作用下的移动,抵抗力抗力会出现波动,因此应力应变曲线出现屈服现象,如下图波浪线部分。
简单的位错能量可以从微观仿真计算得到。但是由于在实际的晶体材料中位错太普遍且杂乱,不同类型的位错带来的应力应变关系的改变也不尽相同。当位不同错交织在一起后,再从微观角度搭建结构计算位错能量变得十分困难,而从统计意义上去估计材料强度可能更加恰当。宏观世界的弹性矩阵~微观的晶格弹性矩阵+mean(位错影响)。
同位错一同出现缺陷的可能有点缺陷,堆垛层错等,这些缺陷共同导致的晶体内部结构对称性的破缺。至此完美晶体渐行渐远。啊?宏观世界要与微观世界脱节了?
晶界
Too yong to simple,其实就弹性来说,位错的影响实在算不上太大。来吧,晶界改变一切。
晶界如下图所示。晶界是结构相同而取向不同晶粒之间的界面。在晶界面上,原子排列从一个取向过渡到另一个取向,故晶界处原子排列处于过渡混乱状态(几到十几个原子厚度)。 晶粒与晶粒之间的接触界面叫做晶界。

为什么好好的一块单晶为什么要变碎了呢?
这我们回要到液态倒推一下。在液体中的原子和分子处于杂乱状态,液态到固态的转变过程中,在液体中存在能量起伏,些许杂乱的微小区域将发生有序排列,变为固体微粒,称为形核(当然也会湮灭消失,重新变回液体)。
当局部区域的液相固相的相变能量大于晶粒界面能(生成)时,晶粒变化变会不断长大,这些不断长大的晶粒是通常遵循核心区域原子排列,所以相互之间并不统一对齐到一个方向,当固相晶粒相互接壤时,不同方向的晶粒便形成了晶界。 (补充:习惯了微观计算的人往往从0K角度思考问题,再尝试在初始结构中附加温度,反应温度对构型与性能的影响。但是对于介观、宏观角度来说,从高温状态变为低温状态是惯有的考虑路线。如,从液态凝固为固态发生一些系列变化。这也是后续系列中,相图和相场的出发点。)
Oh,碎晶粒是正常的,单晶是不正常的。那什么时候倾向于形成多晶,什么时候单晶呢? 在满足充足时间扩散下,这就涉及到晶界能量和构型熵的博弈了。如纯铝的晶粒一般要比纯铁的粗大,基本肉眼可见。
晶粒晶界彻底断绝了宏观性能和微观力学性能的直接联系,首先,如弹性来说,各项异性的晶粒在组合为一块整体的时候,取性能均值,在宏观表现上变现为各向同性。宏观材料上,便可以只能使用弹性常数单个标量体现弹性属性。第二,晶界的高强度界面能,导致材料不容易发生塑性形变,带来了宏观弹性的极大提升,称为晶界强化(细晶强化)。因此材料人员往往通过多种处理方式让晶粒细化。但另一方面,塑性通常会下降。断裂性能上,也不再是单纯的45度切向拉断,穿晶、沿晶断裂各种机制出现。当然也有例外,在高温下,由于晶界位置分子运动加剧(可以当作冰糖化了、变软了),多晶的材料强度反而不如单晶完美结构,这特殊机制促进了研究人员在高温材料的特色研发。
当然,界面不只包括晶界,还包括晶粒内部的亚晶界,摇摆在晶粒内外的栾晶界,以及更高一层的相界面,最外层的表面blablabla。其中比较有特色的栾晶界是指两个晶体(或者一个晶体的两部分)沿着公共晶面构成晶面对称的位向关系。如下图所示。孪晶界面能基本保持了原先结构的对称性,所以能量相对于晶界只有1/10左右。

代码部分:
下面以一个简单的栾晶界展示的案例(本例子实际上也是微观的层错),首先我们先安装必要的软件和定义一个展示用的画图函数。
Looking in indexes: https://pypi.tuna.tsinghua.edu.cn/simple Requirement already satisfied: pymatgen in /opt/mamba/lib/python3.10/site-packages (2023.8.10) Requirement already satisfied: plotly>=4.5.0 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (5.16.1) Requirement already satisfied: palettable>=3.1.1 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (3.3.3) Requirement already satisfied: sympy in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (1.12) Requirement already satisfied: numpy>=1.20.1 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (1.24.1) Requirement already satisfied: tqdm in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (4.64.1) Requirement already satisfied: uncertainties>=3.1.4 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (3.1.7) Requirement already satisfied: spglib>=2.0.2 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (2.0.2) Requirement already satisfied: networkx>=2.2 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (3.1) Requirement already satisfied: tabulate in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (0.9.0) Requirement already satisfied: pandas in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (1.5.2) Requirement already satisfied: requests in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (2.28.1) Requirement already satisfied: pydantic<2.0.0 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (1.10.12) Requirement already satisfied: mp-api>=0.27.3 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (0.34.3) Requirement already satisfied: matplotlib>=1.5 in /opt/mamba/lib/python3.10/site-packages/matplotlib-3.7.1-py3.10-linux-x86_64.egg (from pymatgen) (3.7.1) Requirement already satisfied: pybtex in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (0.24.0) Requirement already satisfied: joblib in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (1.3.2) Requirement already satisfied: monty>=3.0.2 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (2022.9.9) Requirement already satisfied: ruamel.yaml>=0.17.0 in /opt/mamba/lib/python3.10/site-packages (from pymatgen) (0.17.32) Requirement already satisfied: scipy>=1.5.0 in /root/.local/lib/python3.10/site-packages (from pymatgen) (1.11.2) Requirement already satisfied: contourpy>=1.0.1 in /opt/mamba/lib/python3.10/site-packages/contourpy-1.0.7-py3.10-linux-x86_64.egg (from matplotlib>=1.5->pymatgen) (1.0.7) Requirement already satisfied: cycler>=0.10 in /opt/mamba/lib/python3.10/site-packages/cycler-0.11.0-py3.10.egg (from matplotlib>=1.5->pymatgen) (0.11.0) Requirement already satisfied: fonttools>=4.22.0 in /opt/mamba/lib/python3.10/site-packages/fonttools-4.39.4-py3.10.egg (from matplotlib>=1.5->pymatgen) (4.39.4) Requirement already satisfied: kiwisolver>=1.0.1 in /opt/mamba/lib/python3.10/site-packages/kiwisolver-1.4.4-py3.10-linux-x86_64.egg (from matplotlib>=1.5->pymatgen) (1.4.4) Requirement already satisfied: packaging>=20.0 in /opt/mamba/lib/python3.10/site-packages (from matplotlib>=1.5->pymatgen) (22.0) Requirement already satisfied: pillow>=6.2.0 in /opt/mamba/lib/python3.10/site-packages/Pillow-9.5.0-py3.10-linux-x86_64.egg (from matplotlib>=1.5->pymatgen) (9.5.0) Requirement already satisfied: pyparsing>=2.3.1 in /opt/mamba/lib/python3.10/site-packages/pyparsing-3.1.0b2-py3.10.egg (from matplotlib>=1.5->pymatgen) (3.1.0b2) Requirement already satisfied: python-dateutil>=2.7 in /opt/mamba/lib/python3.10/site-packages (from matplotlib>=1.5->pymatgen) (2.8.2) Requirement already satisfied: msgpack in /opt/mamba/lib/python3.10/site-packages (from mp-api>=0.27.3->pymatgen) (1.0.5) Requirement already satisfied: setuptools in /opt/mamba/lib/python3.10/site-packages (from mp-api>=0.27.3->pymatgen) (65.5.0) Requirement already satisfied: typing-extensions>=3.7.4.1 in /opt/mamba/lib/python3.10/site-packages (from mp-api>=0.27.3->pymatgen) (4.7.1) Requirement already satisfied: emmet-core>=0.54.0 in /opt/mamba/lib/python3.10/site-packages (from mp-api>=0.27.3->pymatgen) (0.64.6) Requirement already satisfied: tenacity>=6.2.0 in /opt/mamba/lib/python3.10/site-packages (from plotly>=4.5.0->pymatgen) (8.2.3) Requirement already satisfied: idna<4,>=2.5 in /opt/mamba/lib/python3.10/site-packages (from requests->pymatgen) (3.4) Requirement already satisfied: certifi>=2017.4.17 in /opt/mamba/lib/python3.10/site-packages (from requests->pymatgen) (2022.9.24) Requirement already satisfied: urllib3<1.27,>=1.21.1 in /opt/mamba/lib/python3.10/site-packages (from requests->pymatgen) (1.26.11) Requirement already satisfied: charset-normalizer<3,>=2 in /opt/mamba/lib/python3.10/site-packages (from requests->pymatgen) (2.1.1) Requirement already satisfied: ruamel.yaml.clib>=0.2.7 in /opt/mamba/lib/python3.10/site-packages (from ruamel.yaml>=0.17.0->pymatgen) (0.2.7) Requirement already satisfied: future in /opt/mamba/lib/python3.10/site-packages (from uncertainties>=3.1.4->pymatgen) (0.18.3) Requirement already satisfied: pytz>=2020.1 in /opt/mamba/lib/python3.10/site-packages (from pandas->pymatgen) (2022.7) Requirement already satisfied: PyYAML>=3.01 in /opt/mamba/lib/python3.10/site-packages (from pybtex->pymatgen) (6.0) Requirement already satisfied: latexcodec>=1.0.4 in /opt/mamba/lib/python3.10/site-packages (from pybtex->pymatgen) (2.0.1) Requirement already satisfied: six in /opt/mamba/lib/python3.10/site-packages (from pybtex->pymatgen) (1.16.0) Requirement already satisfied: mpmath>=0.19 in /opt/mamba/lib/python3.10/site-packages (from sympy->pymatgen) (1.3.0) WARNING: Running pip as the 'root' user can result in broken permissions and conflicting behaviour with the system package manager. It is recommended to use a virtual environment instead: https://pip.pypa.io/warnings/venv Looking in indexes: https://pypi.tuna.tsinghua.edu.cn/simple Requirement already satisfied: ase in /opt/mamba/lib/python3.10/site-packages (3.23.0b1) Requirement already satisfied: scipy>=1.3.1 in /root/.local/lib/python3.10/site-packages (from ase) (1.11.2) Requirement already satisfied: numpy>=1.17.0 in /opt/mamba/lib/python3.10/site-packages (from ase) (1.24.1) Requirement already satisfied: matplotlib>=3.1.0 in /opt/mamba/lib/python3.10/site-packages/matplotlib-3.7.1-py3.10-linux-x86_64.egg (from ase) (3.7.1) Requirement already satisfied: contourpy>=1.0.1 in /opt/mamba/lib/python3.10/site-packages/contourpy-1.0.7-py3.10-linux-x86_64.egg (from matplotlib>=3.1.0->ase) (1.0.7) Requirement already satisfied: cycler>=0.10 in /opt/mamba/lib/python3.10/site-packages/cycler-0.11.0-py3.10.egg (from matplotlib>=3.1.0->ase) (0.11.0) Requirement already satisfied: fonttools>=4.22.0 in /opt/mamba/lib/python3.10/site-packages/fonttools-4.39.4-py3.10.egg (from matplotlib>=3.1.0->ase) (4.39.4) Requirement already satisfied: kiwisolver>=1.0.1 in /opt/mamba/lib/python3.10/site-packages/kiwisolver-1.4.4-py3.10-linux-x86_64.egg (from matplotlib>=3.1.0->ase) (1.4.4) Requirement already satisfied: packaging>=20.0 in /opt/mamba/lib/python3.10/site-packages (from matplotlib>=3.1.0->ase) (22.0) Requirement already satisfied: pillow>=6.2.0 in /opt/mamba/lib/python3.10/site-packages/Pillow-9.5.0-py3.10-linux-x86_64.egg (from matplotlib>=3.1.0->ase) (9.5.0) Requirement already satisfied: pyparsing>=2.3.1 in /opt/mamba/lib/python3.10/site-packages/pyparsing-3.1.0b2-py3.10.egg (from matplotlib>=3.1.0->ase) (3.1.0b2) Requirement already satisfied: python-dateutil>=2.7 in /opt/mamba/lib/python3.10/site-packages (from matplotlib>=3.1.0->ase) (2.8.2) Requirement already satisfied: six>=1.5 in /opt/mamba/lib/python3.10/site-packages (from python-dateutil>=2.7->matplotlib>=3.1.0->ase) (1.16.0) WARNING: Running pip as the 'root' user can result in broken permissions and conflicting behaviour with the system package manager. It is recommended to use a virtual environment instead: https://pip.pypa.io/warnings/venv
Co2
首先,从pymatgen现在Co金属的结构文件"mp-54",保存为“base.cif”。pymatgen 输入的API key可以从material project官网申请。Co是典型的HCP堆垛结构,即沿着[110]面,A、B两中排列方式相互插空排列。(A-B-A-B-A-B)
我们将某一层的Co原子替换,构造z轴晶面对称的结构。如下图所示。经过替换后,可以看出,在[110]晶面,中间某一层以fcc堆垛。
Co80 Co80


未完待续ing
下节:相,析出,固溶,相图计算







